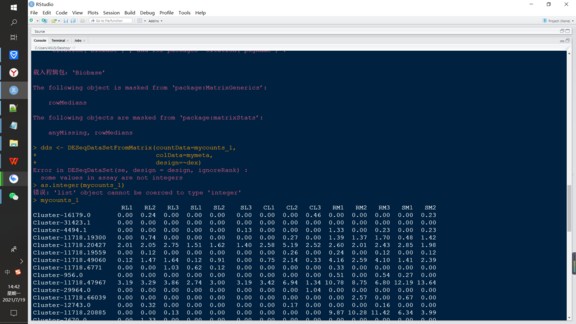
是的 , DESeq2要求输入的是readcount 值 为整数,你这数据不是整数不能输入;
各位大佬好!我在筛选一批转录组数据里的差异基因时,当进行到构建dds对象的时候,r语言出现以下报错,说“一些值不是整数”,当我上网查这个错误的时候,有人说是“数据已进行归一化,无法进行DESeq2分析”,请问各位大佬,还有什么解决方法嘛??谢谢谢谢
2 个回答
- 2 关注
- 0 收藏,4388 浏览
- 学而时嘻嘻 提出于 2021-07-19 14:44
相似问题
- 差异基因表达分析 1 回答
- 老师,请帮忙,感谢。结果有参转录组5.deg报错。 2 回答
- 两组数据每组2个样品,对比差异基因,应该用什么方法? 1 回答
- 关于课程中TCGA差异基因表达代码-问题 1 回答
- 转录组差异分析里GO注释的图怎么看的? 1 回答