如果是我们公司做的项目,有问题可以找对应的销售,他们会给您详细的解答。
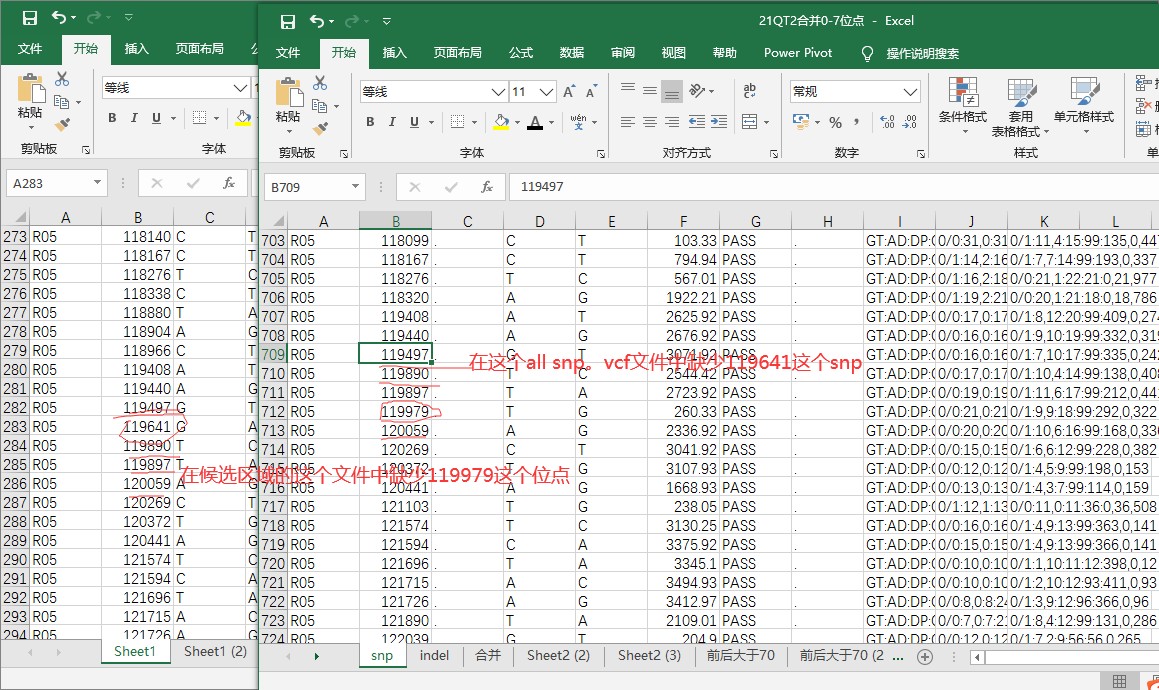
在BSA分析结果给的候选区域文件quantile99_SNPs_in_region_gene_anno中,发现与all_sample_snp.vcf对比后,发现好多SNP位点没有,请问是什么原因?
在BSA分析结果中,对候选区域文件quantile99_SNPs_in_region_gene_anno的SNP位点进行查看并与all_sample_snp.vcf对比发现在这个候选区域中,有些位点没有,这是什么原因? 候选区域及候选区域基因注释的文件中,如snp位点142198是一个在父母本中纯合的位点,但是在候选区域中的位点中却没有这个位点,请问是什么原因(这个BSA分析是在你们公司做的),为什么会被过滤掉?同样在候选区域文件quantile99_SNPs_in_region_gene_ann中的一些位点在all_sample_snp.vcf中也没有?我的理解不应该是候选区域文件中应该是包括这段区域里面所有的snp吗?
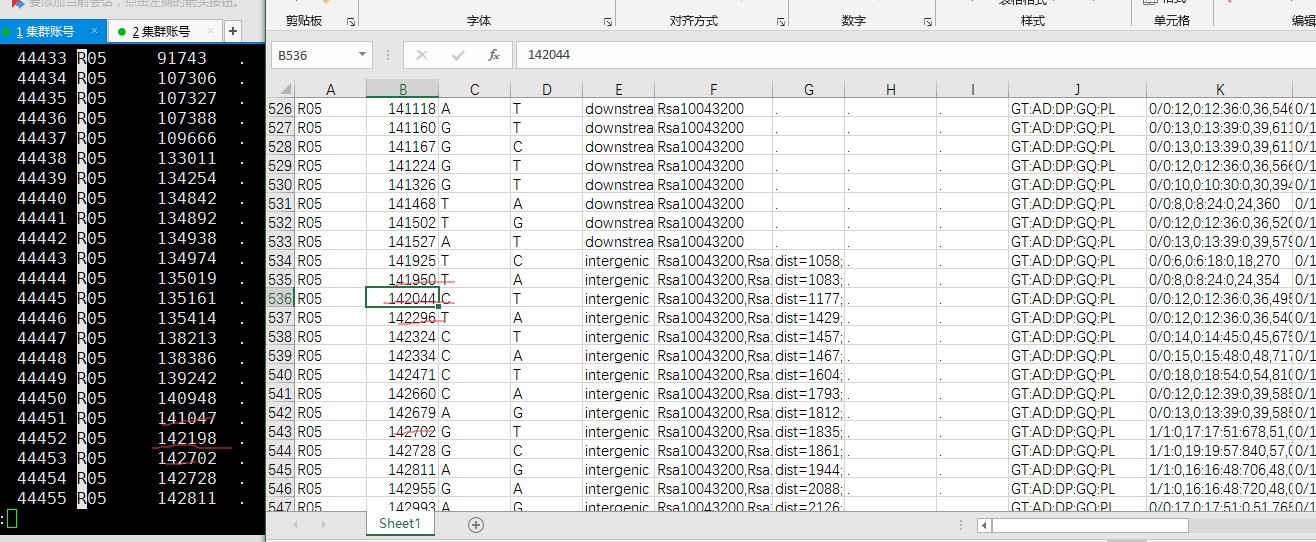
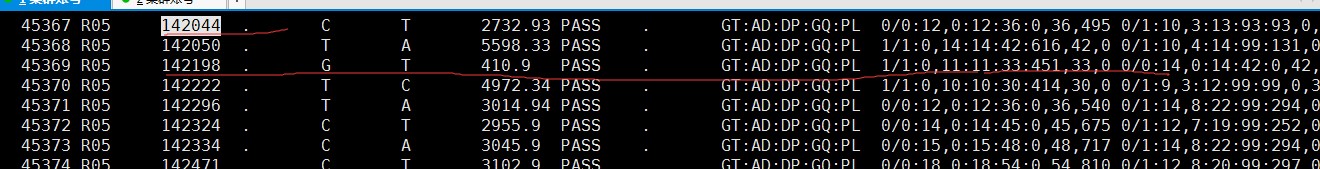
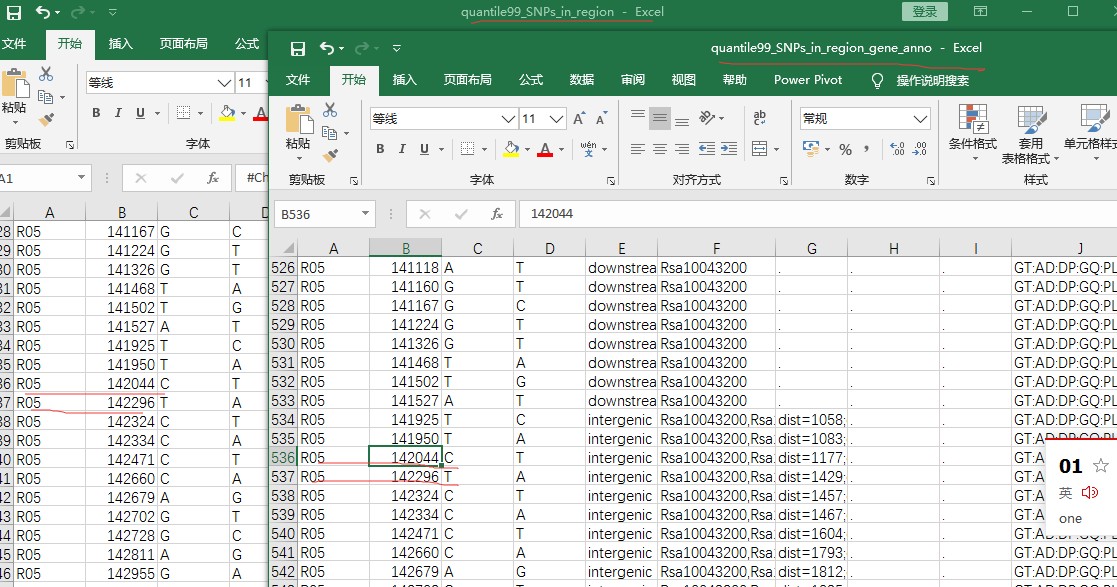
下面的这两个文件分别是从候选区域和所有snp位点的文件中红截取的相同区域
请先 登录 后评论
1 个回答
- 1 关注
- 0 收藏,2336 浏览
- 杨辉 提出于 2021-08-04 17:19
