首先 这个脚本是物种分类,这里的相似性是序列与库中的序列的相似性;
我们通常说的聚类相似性97%得到OTU,那是序列聚类,你需要分清楚。
我怀疑你是不是搞错了
如题,请教。如何修改assign命令下blast的比对阈值?
qiime1的assign_taxonomy.py命令可以用uclust, blast, sortmerna, rdp等方法比对,同时可以通过修改一些比对的阈值来设置比对的‘门槛’。
但我发现,使用blast方法比对时的阈值,官网上提供的可修改的阈值参数只有一个e value,即使用 -e 命令修改,但是其它blast参数是没法设置的。
然而像我们手动在NCBI进行blast比对时,结果里有除了e value以外,还有bit score, identity等指标可以参考。虽然qiime1的assign有一个配套的 --similarity(比对时的相似度) 命令,但提示仅供uclust和sortmerna方法使用。
不知-m blast的其它参数的修改是在qiime1里根本就没法实现?还是官网上没有交代?
对此很困惑,因为感觉这应该是个很基本的需求,不能改就太离谱了。。。而且,blast比对输出结果的.log文件提供的信息里,会显示一个 默认值min percent identity 90.0(90%)(如图),然而这个值是不能修改或设置的吗? 我现在需要在identity 97%的条件下blast,但不知如何实现。。。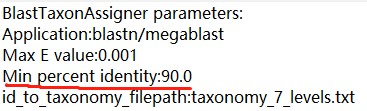
老师您好,感谢回答。抱歉我在问题中可能没有描述清,有些术语可能用的不够专业。
具体情况是:我现在已经得到了OTU的代表序列和OTU table,然后打算给OTU代表序列使用assign命令做物种分类和注释。
我是想用blast方法(即assign_taxonomy.py -m blast -r 参比序列数据库 -t 参比序列id)进行物种分类后,先把非目标序列从OTU表格中剔除掉,然后再做后续分析。
目前这个assign_taxonomy.py操作在执行上没有问题。但就是发现,通过blast方法比对-分类时,由于qiime默认的blast相似度阈值比较低,导致很多不是目标微生物的片段(因有不少非特异性扩增)也可以和参比序列数据库比对上,所以我无法在分类注释结果中把它们和真正的目的片段区分开。
这个我已初步试过,即通过调整blast的e value(即在assign命令后加-e命令,如-e 1e-50)后,可以大幅改善注释结果,很多非目标序列的注释结果都会变成no blast hit,不会再被assign一个分类结果。
但是现在的问题是,我发现仅靠修改e值是不够的。我后续手动在NCBI上用blastn检查了一些调整e值后可以注释上的序列,发现很多序列e值虽然比较好,但是blast score和identity什么的非常低,说明它们可能仍然不是目标序列。
所以现在想通过继续提高其它blast的阈值来实现。但不知具体改如何操作,不知assign命令下的blast方法,除了e value外,还有没有办法可以调整其它阈值。。。?
例如,我看一些文献中,在用blast给序列分类注释时,除了会把e值设为1e-50外,还设置了如blast score > 200, identity >97%之类的,这些在qiime里该如何实现?
您好,请问您的blast的过程中有没有遇到burrito.util.ApplicationNotFoundError: Cannot find formatdb. Is it installed? Is it in your path?错误,虽然我已经安装blast,也用命令进行了blast建库,但还是会报错