5 系统发育树:基因结构比较散,长度差异大,如何绘制满意的系统发育树?
老师,您好!我目前在学习基因家族分析,在构建进化树时一直无法获得一个结果较好的树
猜想主要原因是由于Alignment时本来就比得不齐,比对结果如下
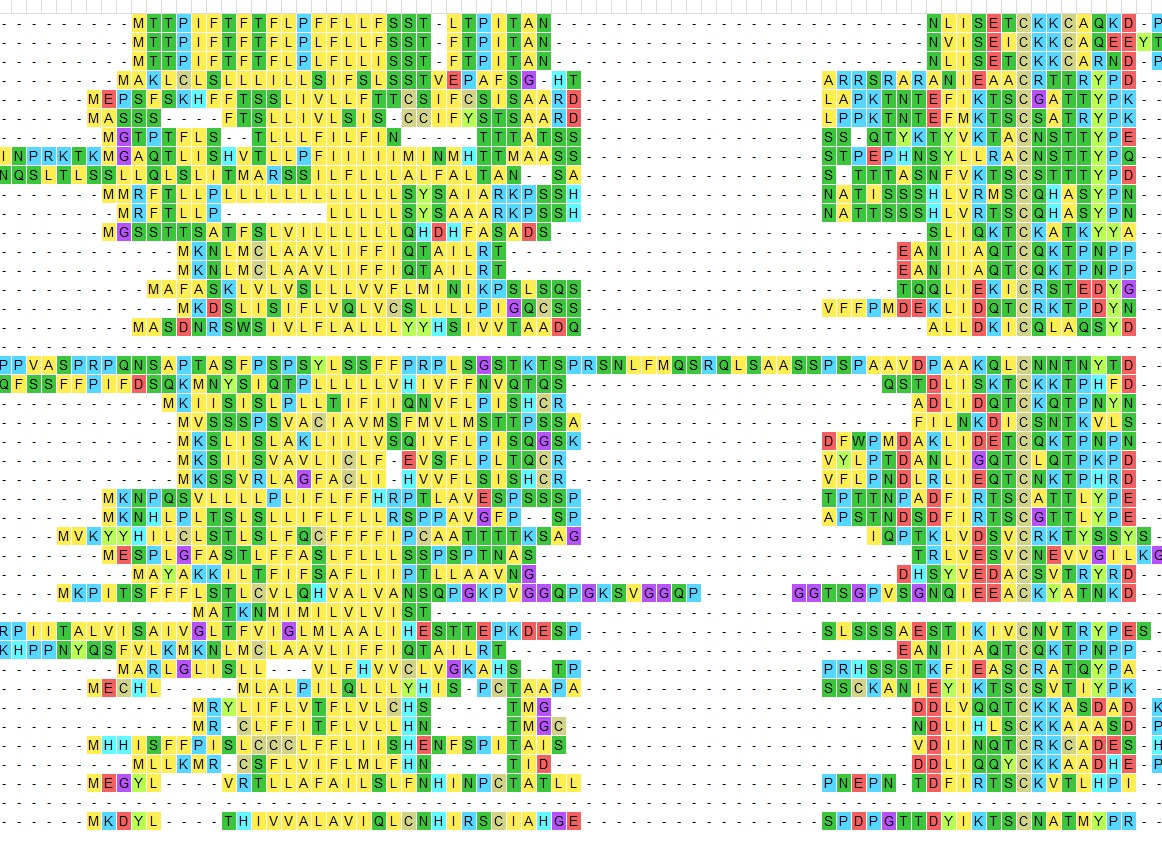 删除空白gap后:
删除空白gap后: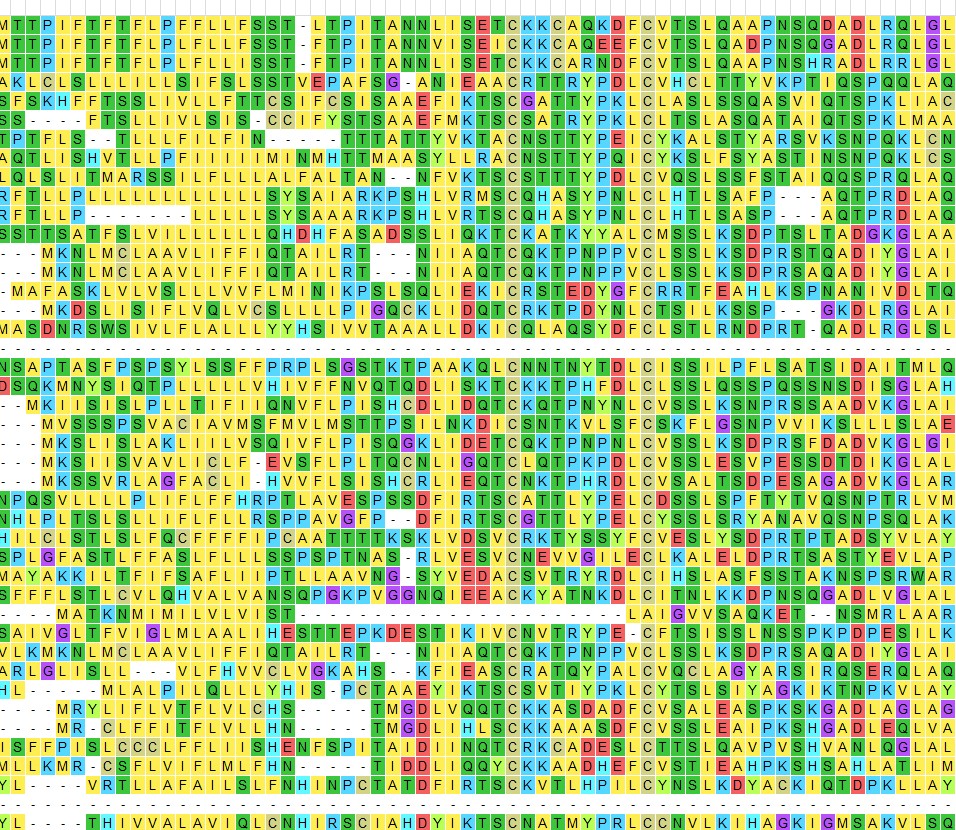
导出mega formate打开:比对结果如下,只有一列保守的区域,显然不符合一个基因家族的特性

使用的软件是MEGA,采用了clustalw和Muscle两种方法,比出的结果都不尽人意。
于是绘制了该基因家族的motif结构图:
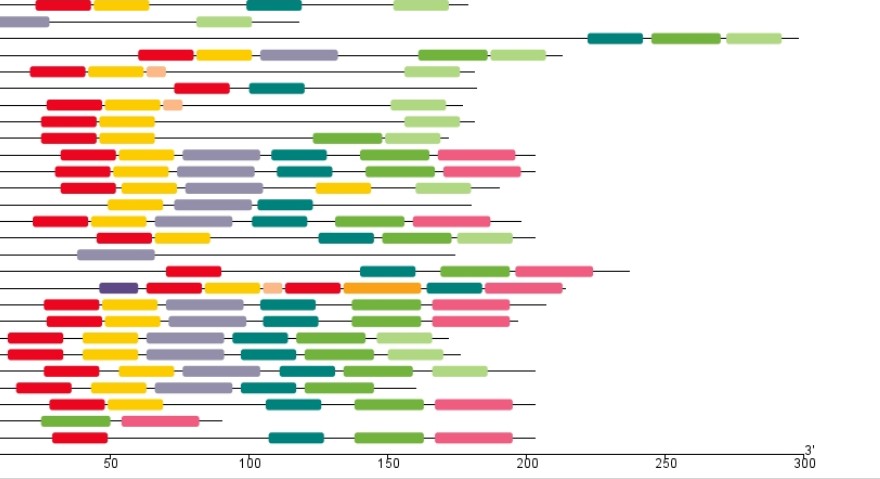 可以看出该基因家族的基因长度差异较大,motif的分布也比较散,
可以看出该基因家族的基因长度差异较大,motif的分布也比较散,
请教一下老师,在这种情况下,我该如何取舍以获得一个良好的系统发育树呢?
是该舍弃掉那些太长或太短的基因吗?可是他们又的确是含有保守结构域的呀?
还是需要手动修正一下序列比对呢?
还望老师不吝赐教,万分感谢!
请先 登录 后评论
最佳答案 2018-10-27 21:00
应该是序列差异太大,可以删除一些大的gap,或者删除一些差异大的基因:
https://www.omicsclass.com/article/221
如果基因全长序列差异太大,建议截取结构域所在序列构建进化树;
如果要编辑MEGA输出的多序列比对结果建议用genedoc:https://www.omicsclass.com/article/502
请先 登录 后评论
如果觉得我的回答对您有用,请随意打赏。你的支持将鼓励我继续创作!

- 2 关注
- 2 收藏,12781 浏览
- 小鹿kity 提出于 2018-10-24 23:20