老师,请教一个问题:
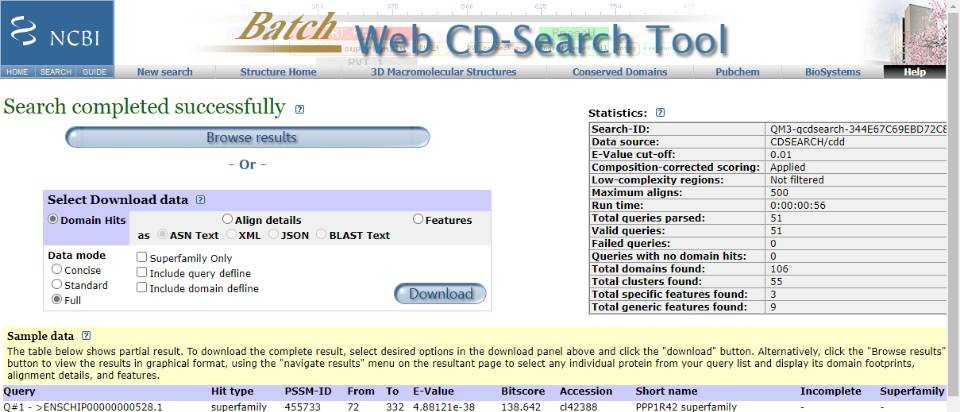
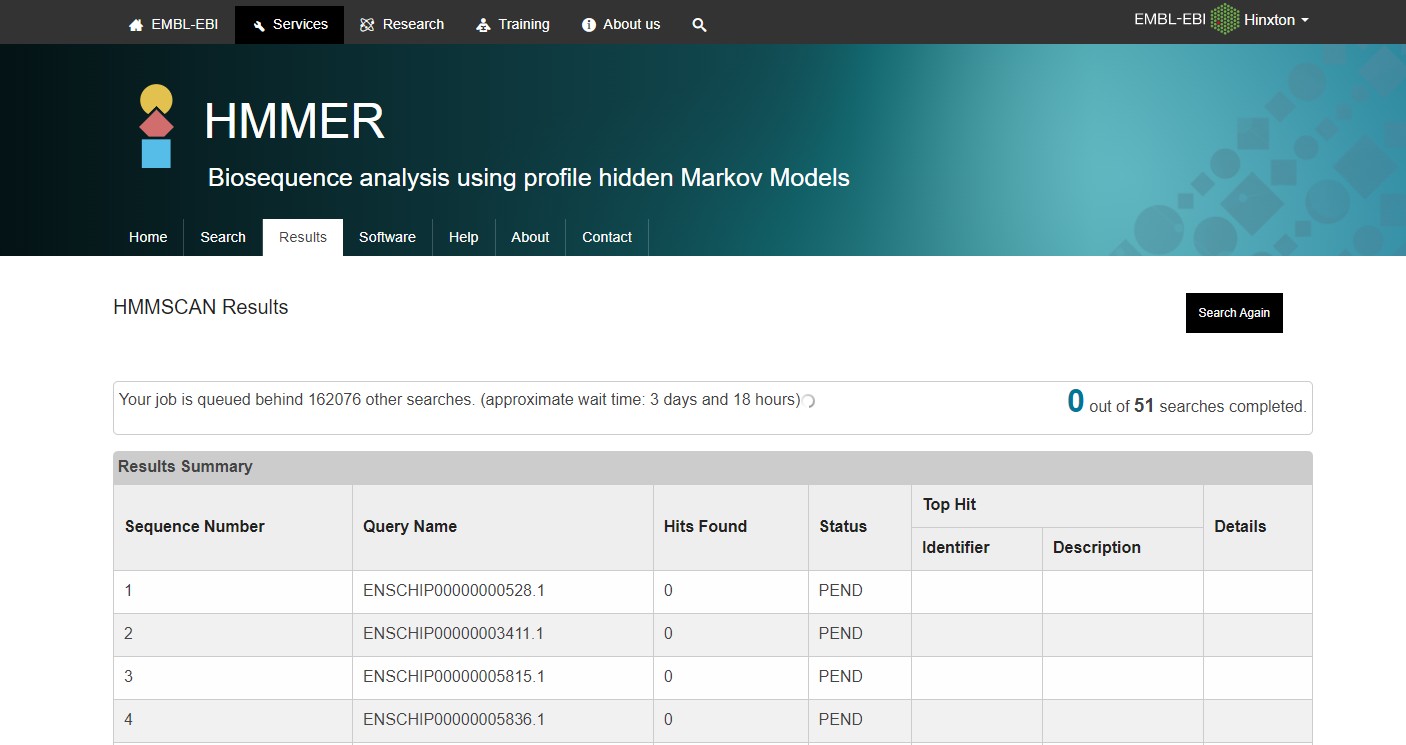
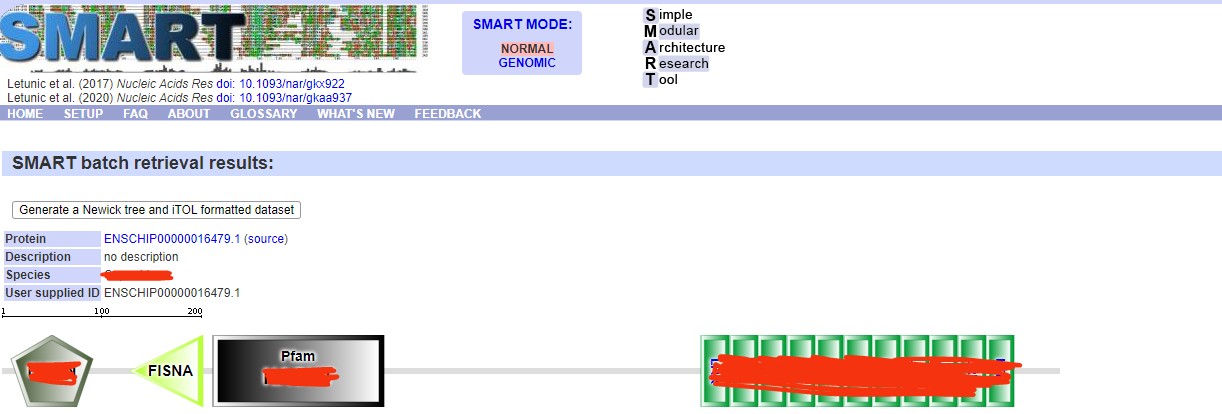
(1)我通过基因家族分析课程19课时的方法,从NCBI下载已知Human 目标基因蛋白序列,获得了XXX.fa 文件。提交到了NCBI-CD-search,获得了如下结果(图一);提交到Pfam数据库,获得了图二暂未出结果(图二);提交到SMART数据库,得到了图三所示结果(图三)。
图一

图二
 图三
图三
在CD-search 里面,Incomplete出现N/C/NC端不完整的片段,是直接从XXXX.fa里面删除整条ID序列吗? 完成三大数据库的比对后,在hmmsearch第一次搜素结构域时,我的如何对接上?