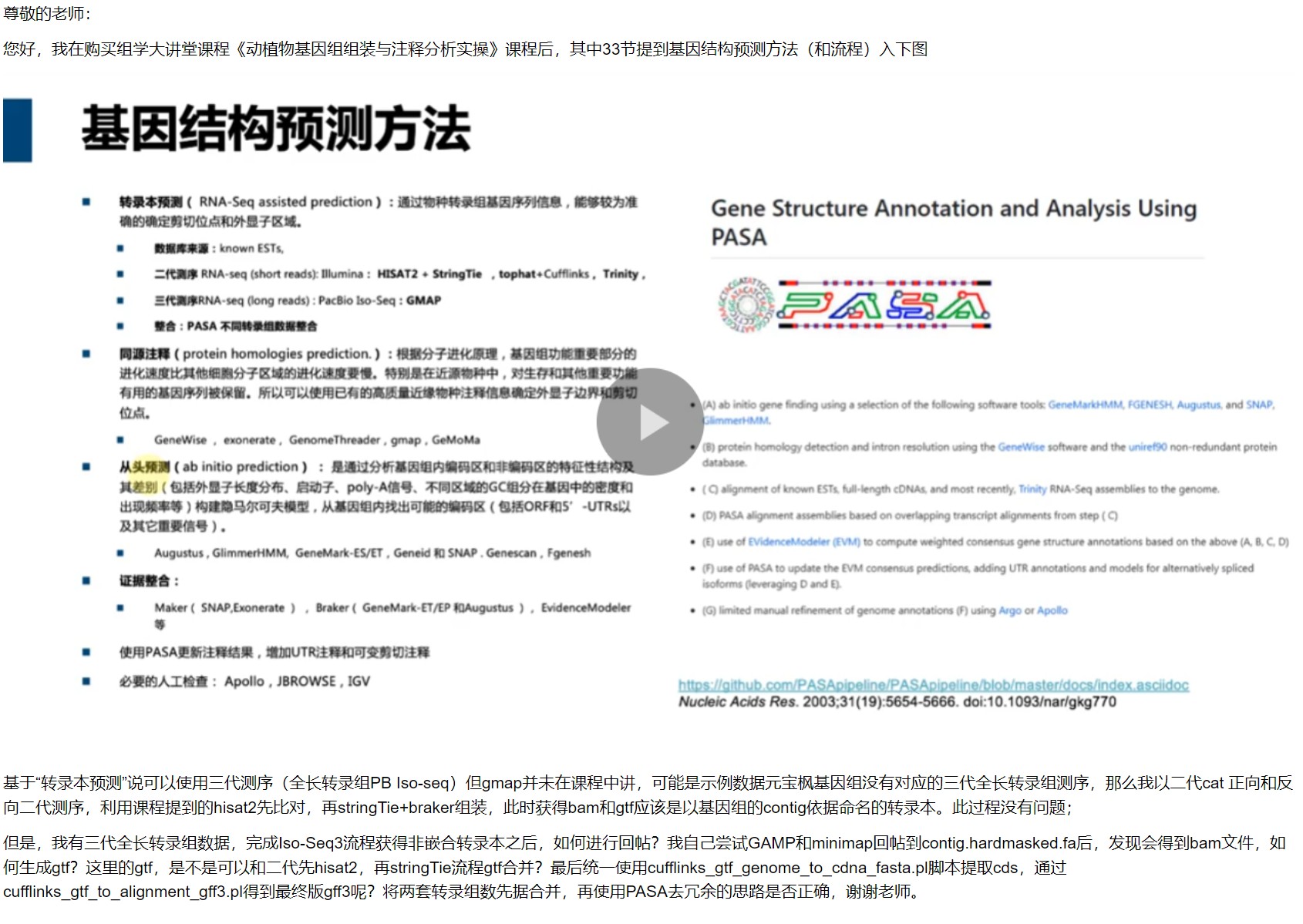
三代的数据可以用pacbio的isoseq工具得到转录本序列,用cdhit去冗余之后的序列输入PASA就行;
如果觉得三代的质量好就设置这些ID到:FL_accs.txt
注意:gmap和blat,minimap的功能一样。我们的基因注释docker镜像中PASA用gmap比对可能得不到结果,所有没有设置,用minimap和lbat效果一样的
参考:
https://github.com/PASApipeline/PASApipeline/issues/131
https://www.frontiersin.org/articles/10.3389/fgene.2021.825742/full#h3
https://academic.oup.com/hr/article/doi/10.1038/s41438-021-00476-4/6446661