1. 我们的deep TE用的是自带的库预测分类,方法不一样;
2.
第99行和第204行的区别:运行RepeatMasker结果文件的不同
第99行:生成的contig.fa.masked文件,其中的重复序列是用N表示
参数不一样,xsmall 表示小写ATCG为repeat soft mask
第204行:同样生成contig,fa.masked,其中的重复序列用小写字母表示。
两者不同,原因是什么?
3.
最后是重复序列同源蛋白预测:这个是重复序列注释流程:
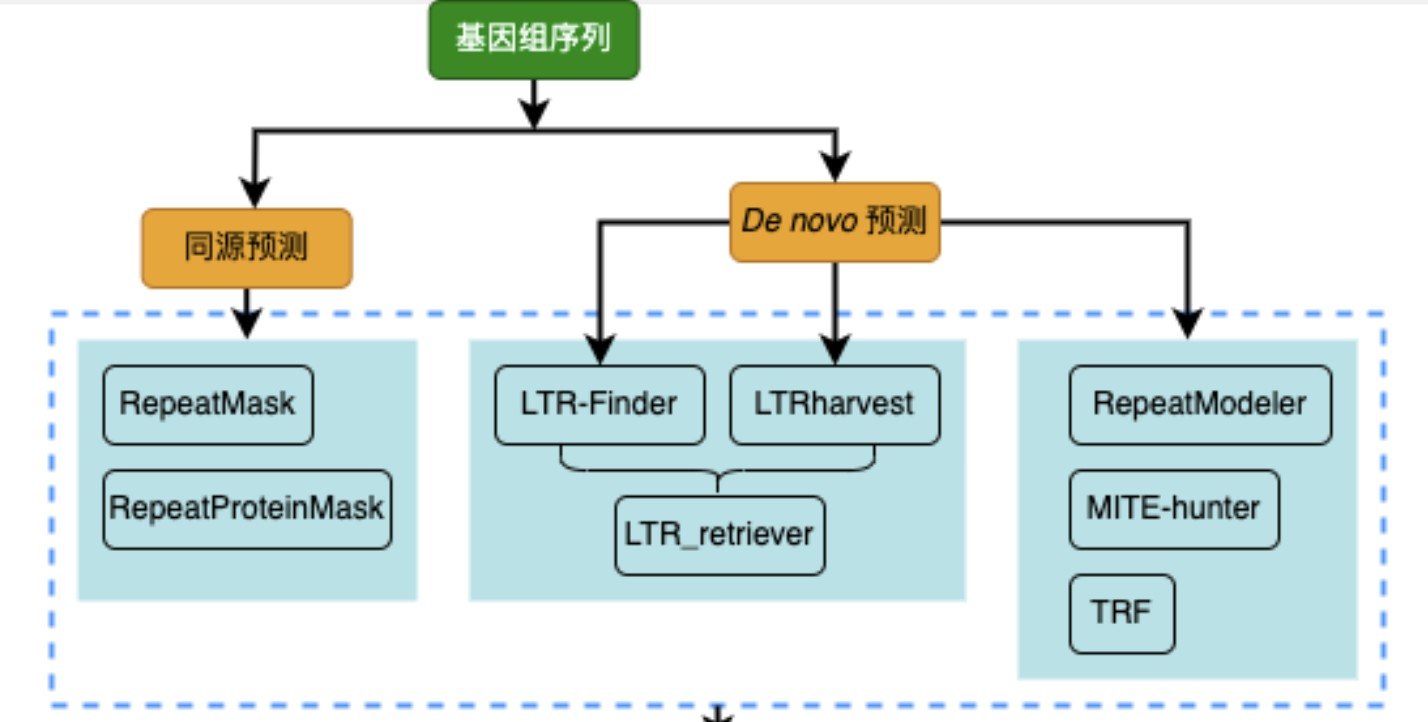
动植物基因组组装课程,01.Repeat.sh中有几个问题:
1、 第175行:#deepTE 分类
根据https://www.jianshu.com/p/85e54962e2bc一文介绍,先用DeepTE_domain.py对保守domain进行鉴定,再用DeepTE.py借助上一步的保守domain信息分类TE。
课程中没有对保守结构域进行鉴定,直接进行TE分类,原因是什么?没有必要?
175行截图:
https://www.jianshu.com/p/85e54962e2bc:网页截图

2、第99行和第204行的区别:运行RepeatMasker结果文件的不同
第99行:生成的contig.fa.masked文件,其中的重复序列是用N表示
第204行:同样生成contig,fa.masked,其中的重复序列用小写字母表示。
两者不同,原因是什么?


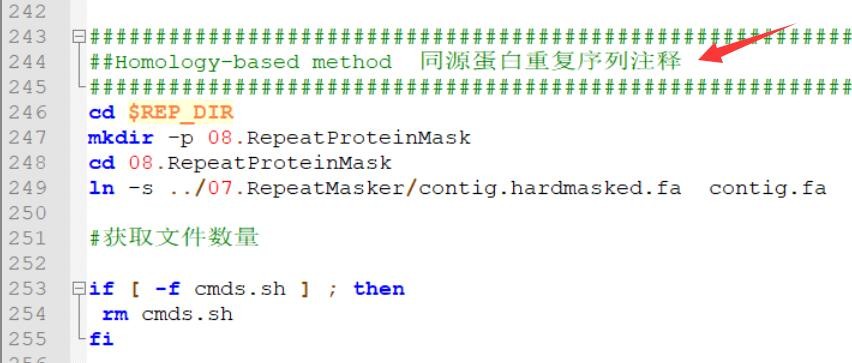
3、第244行:同源蛋白重复序列注释,不太理解这个步骤的目的是啥?请老师详细解释,谢谢!
1. 我们的deep TE用的是自带的库预测分类,方法不一样;
2.
第99行和第204行的区别:运行RepeatMasker结果文件的不同
第99行:生成的contig.fa.masked文件,其中的重复序列是用N表示
参数不一样,xsmall 表示小写ATCG为repeat soft mask
第204行:同样生成contig,fa.masked,其中的重复序列用小写字母表示。
两者不同,原因是什么?
3.
最后是重复序列同源蛋白预测:这个是重复序列注释流程:

如果觉得我的回答对您有用,请随意打赏。你的支持将鼓励我继续创作!
