这个不好判断,需要看具体的情况
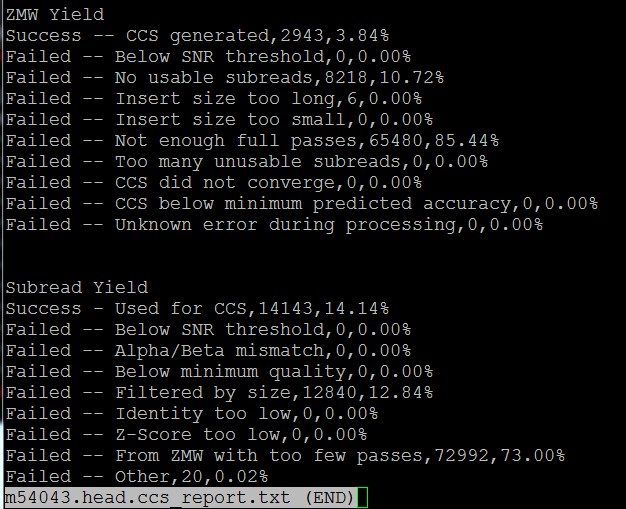
1. 先检查一下第一步ccs 生成的文件,里面有个report 文件吧,看看统计信息。
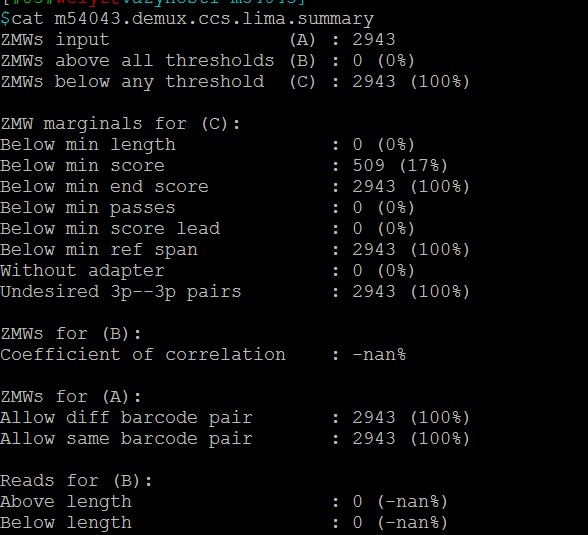
2. 再检查一下第二步lima 里面有个summary 文件。
可以截个图过来看看。
在用IsoSeq3分析时
1)ccs --minLength=200 --minReadScore=0.75 --minPredictedAccuracy=0.8 --numThreads=20 --noPolish --minPasses=1 --reportFile=test.ccs_report.txt test.subreads.bam test.ccs.bam
2)lima test.ccs.bam adapter.fasta test.demux.ccs.bam --isoseq --no-pbi
在第二步生成的结果没有test.demux.ccs.bam,但程序也没有报错,请问会有什么原因造成??
好的 ~~老师
老师还有个问题,我看不懂这些文件中每个统计的含义,请问下可以在那里找到这些文件的结果说明文档吗?
下面是CCS report 截图
 lima的summary文件
lima的summary文件