基因家族分析作为低投入高产出的发文思路,受到广泛关注,分析的人也越来越多,我们也对基因家族分析课程针对之前学员经常遇到的问题进行了更新:
解决基因ID不对应的问题
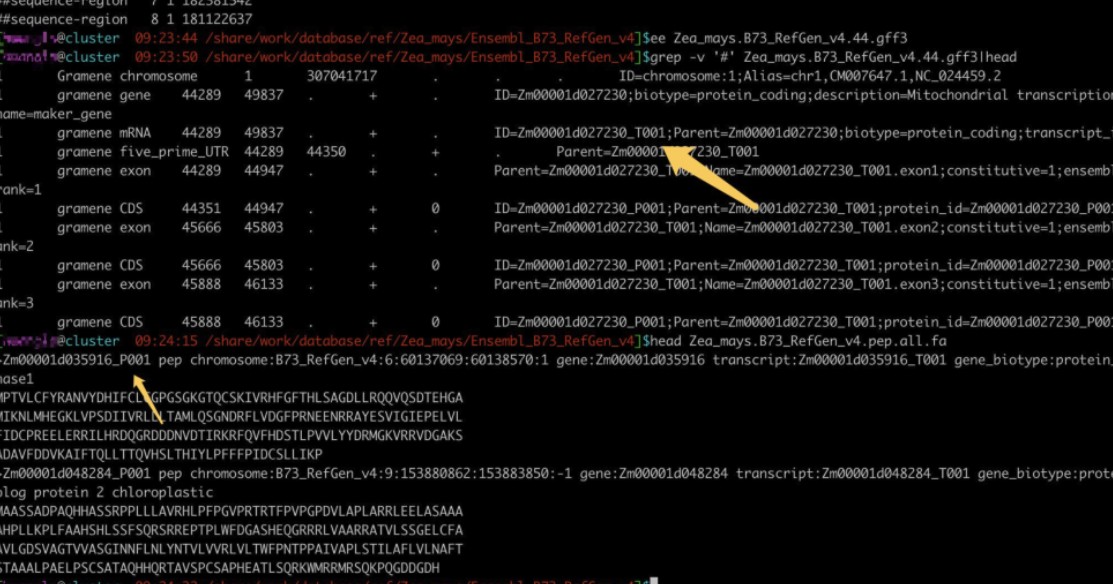
例如上面的玉米基因组,序列中的蛋白ID与GFF中的m RNA ID不对应导致出错,我们使用AGAT自动提取基因组中的cds,可以使GFF与蛋白ID保持一致,为后续的分析扫清障碍。
解决进化树构建序列截取问题
采用 trimAl 自动修改,保留序列中保守的区域用于后续进化树构建。
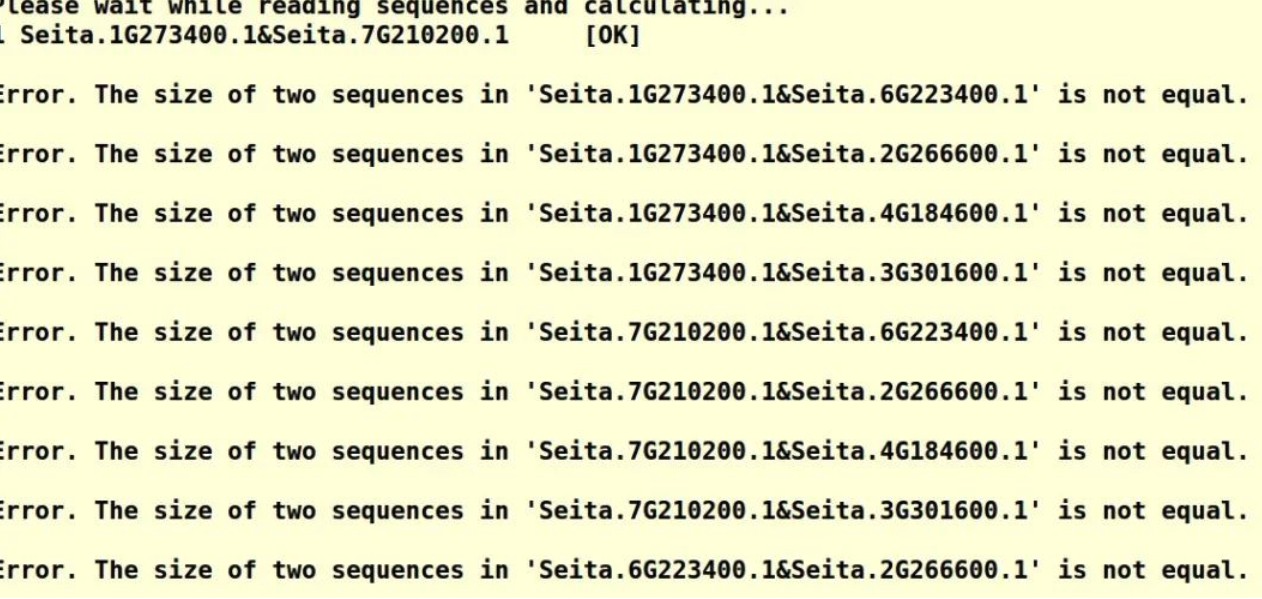
复制基因kaks分析批量计算
采用Para AT,自动批量比对,由蛋白比对再翻译成cds比对,避免比对不按照密码子比对。
4.其他:
流程重新优化,基因家族高分文章思路,分析思路更清晰简洁高效等,分析软件也得到更新,因此我们更新了基因家族镜像2.0,下载使用方法:
docker pull omicsclass/gene-family:v2.0docker run -it --rm omicsclass/gene-family:v2.0
课程观看链接:https://bdtcd.xet.tech/s/1BAqPp
参考文献:
Zhang, Z., Xiao, J., Wu, J., Zhang, H., Liu, G., Wang, X. and Dai, L. (2012) ParaAT: A parallel tool for constructing multiple protein-coding DNA alignments, Biochem Biophys Res Commun, 419(4):779-7
trimAl: a tool for automated alignment trimming in large-scale phylogenetic analyses.Salvador Capella-Gutierrez; Jose M. Silla-Martinez; Toni Gabaldon. Bioinformatics 2009 25: 1972-1973.