这个需要测试查看你的输入文件是不有问题;光从你的描述我也不好推断原因;
这些异常的基因都去看看删除一下试试,还有就是文件不要在windows下用记事本修改导致文件兼容问题;编辑用专业的工具并设置:https://www.omicsclass.com/article/395
老师好,我写了sh后台运行ParaAT 多序列比对
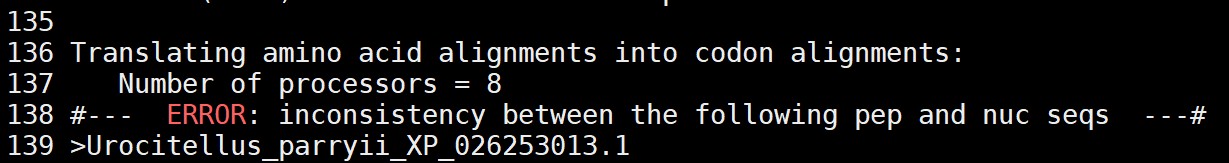
查看运行完成的日志文件时,一共有148个这样的报错情况:
在将氨基酸序列(pep)转换为核酸序列(nuc)时发现了不一致性
我查找了其中三个ID对应的cds和pep序列,碱基/氨基酸个数为:cds=pep*3+3,是3倍关系
——————
并且继续用这个结果进行下一步的合并分析时 #合并成supergene
seqkit concat ./aln_out/*.pep_aln.renamed > single_copy.pep_msa.fasta
输出的结果是空的……
用蛋白质或cds比对结果构建进化树都出现这个情况
——————
前面都是按照课程的脚本跑的
请问该怎么处理这个报错呢?