GATK做vcf合并的模块应该是CombineVariants吧,具体你可以看一下这篇文章:https://www.omicsclass.com/article/1732,里面有写如何做多个vcf的合并
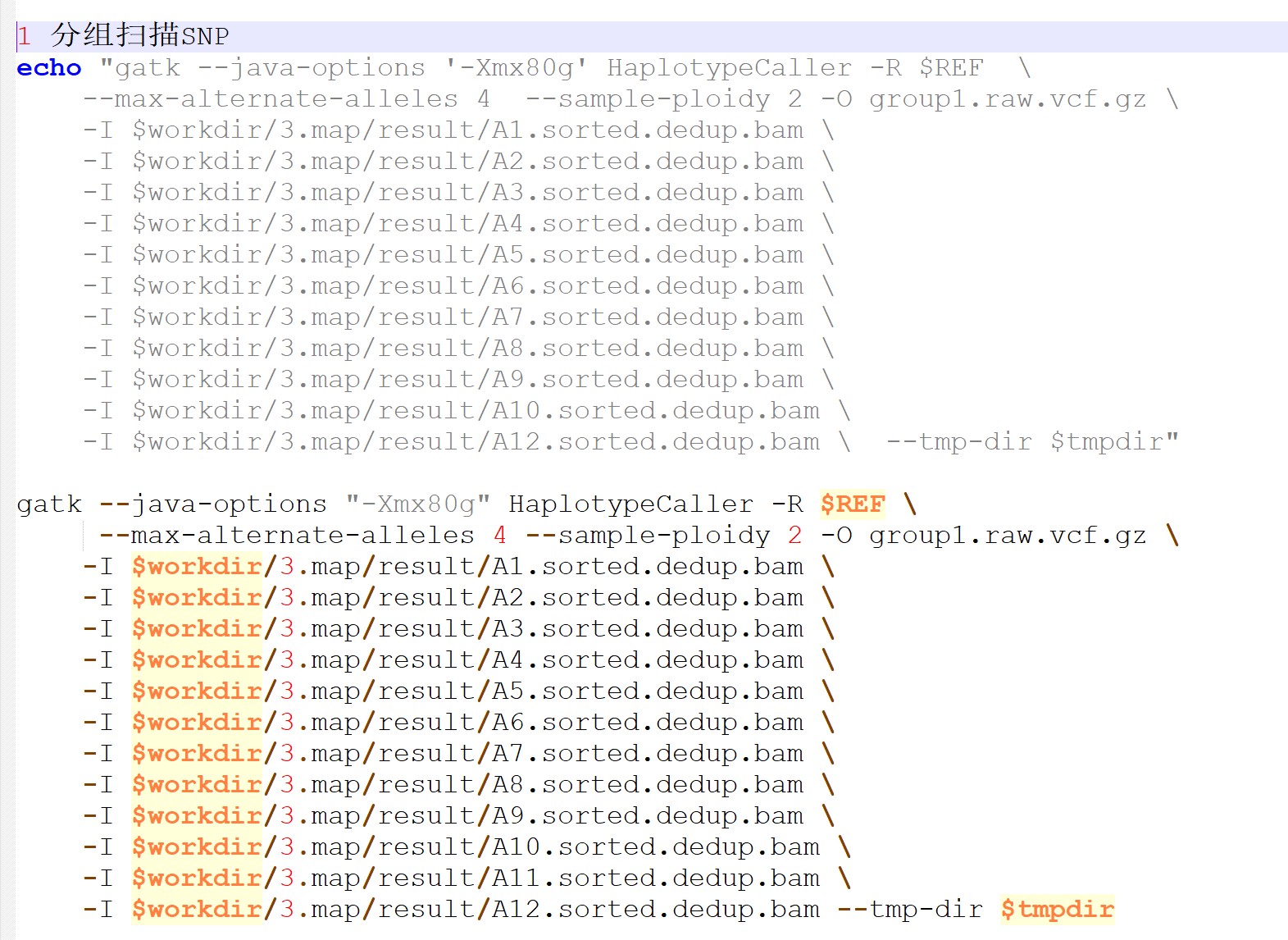
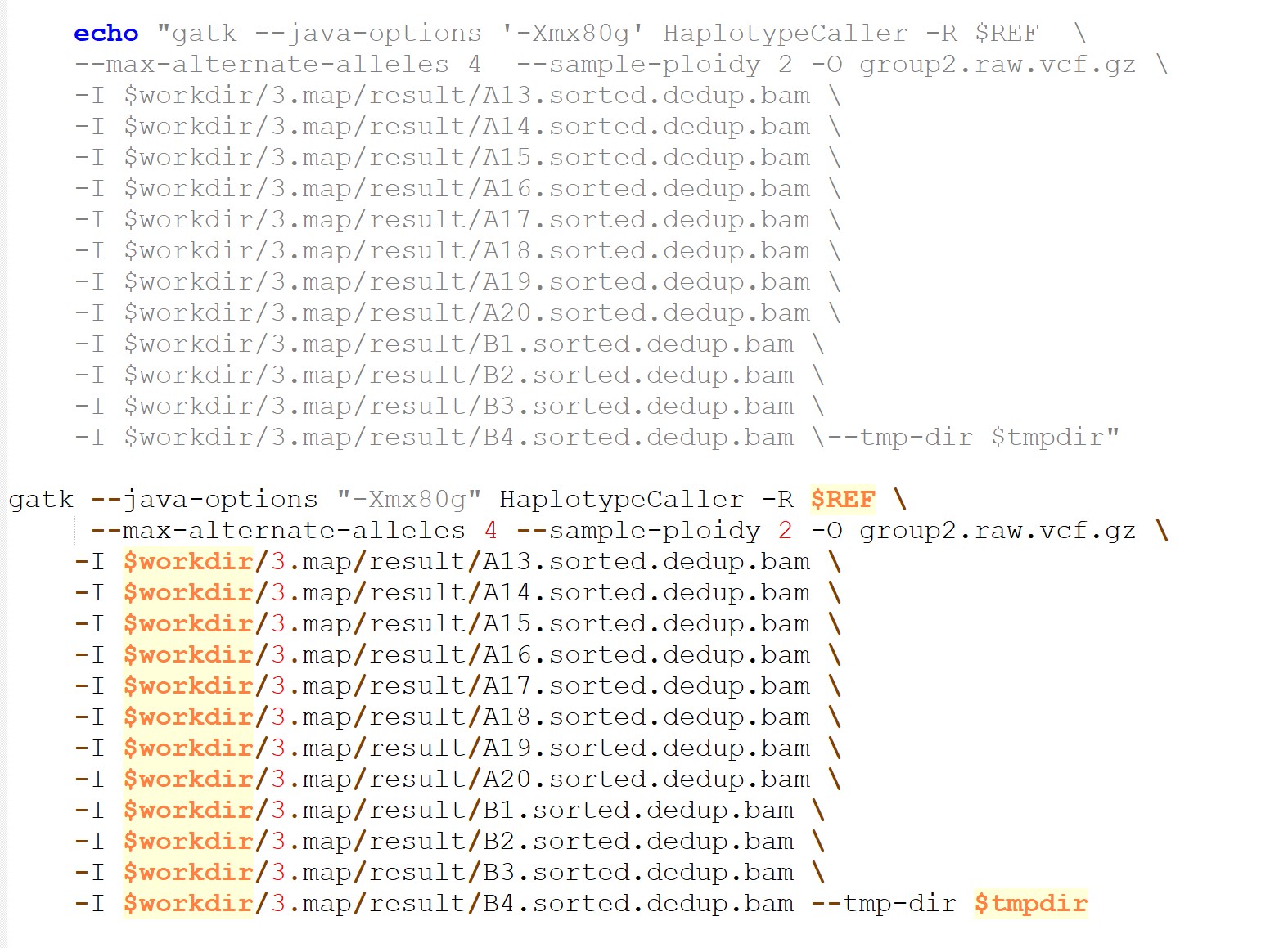
老师,就是我把重测序个体分为三组进行SNP扫描,然后把三次扫描的结果进行合并,得到最后的扫描结果,可以这样操作吗,,下面是代码, 或者是有没有可以将其分开进行snp扫描 然后合并的其他方法,还请老师认真帮我解答 谢谢
2 个回答
擅长:重测序,遗传进化,转录组,GWAS
样本多建议每个样本先生成GVCF,再合并。
你贴的haplotypecaller命令生成的是VCF不是GVCF,下面的代码合并会会报错:

大样本 call SNP 生成GVCF这部分内容重测序课程里面有,你看看的:点击学习:https://bdtcd.xetlk.com/s/1VQOjQ
请先 登录 后评论
如果觉得我的回答对您有用,请随意打赏。你的支持将鼓励我继续创作!

- 1 关注
- 0 收藏,762 浏览
- 烟雨易冷 提出于 2024-11-28 08:35