差异比较分组有问题,需要调试代码看看才行;
泛基因组核心基因和可变基因长度比较core_pan_gene_compare.r报错
运行core_pan_gene_compare.r ,报错Error in contrasts<-(*tmp*, value = contr.funs[1 + isOF[nn]]) :
contrasts can be applied only to factors with 2 or more levels
Calls: aov ... model.matrix.terms -> model.matrix.default -> contrasts<-
Execution halted。我在泛基因组分类的时候只分为了3类,core, dispensable和private。是这个原因导致的报错吗?还是其他的原因?如何修改脚本?core_pan_gene_compare.r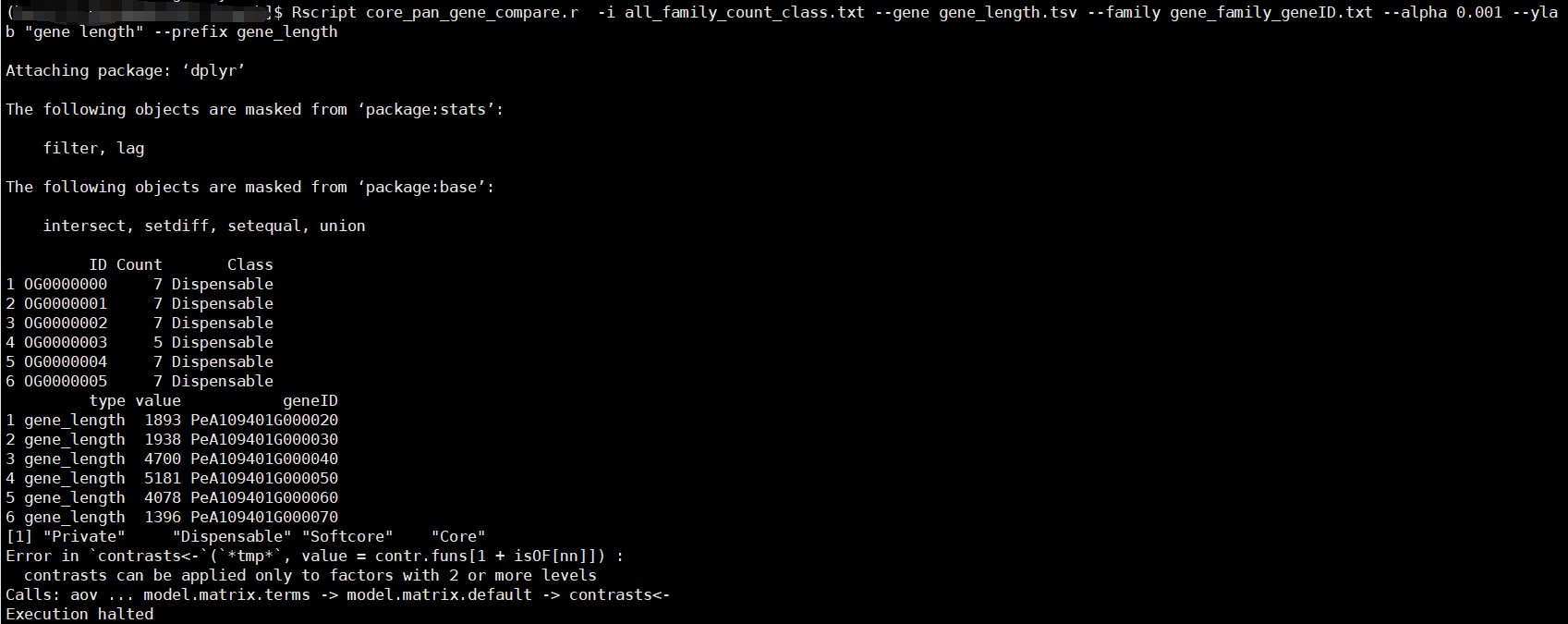
请先 登录 后评论
