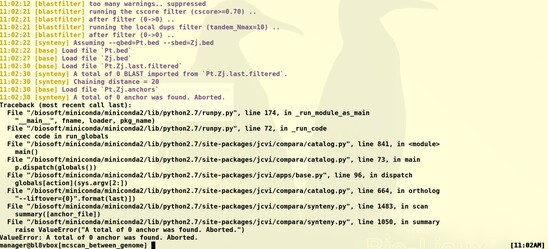
老师好,我在做课时20 比较基因组学分析时 在/biosoft/miniconda/miniconda2/bin/python -m jcvi.compara.catalog ortholog ATH Zj --cscore=0.7这一步出错,换下一个物种生成anchors文件为空,请老师指点
老师好,我在做课时20 比较基因组学分析时 在/biosoft/miniconda/miniconda2/bin/python -m jcvi.compara.catalog ortholog ATH Zj --cscore=0.7这一步出错,换下一个物种生成anchors文件为空,请老师指点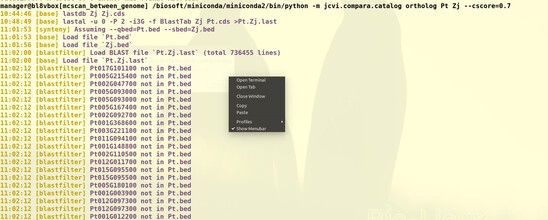

 bed和cds名字也都一样
bed和cds名字也都一样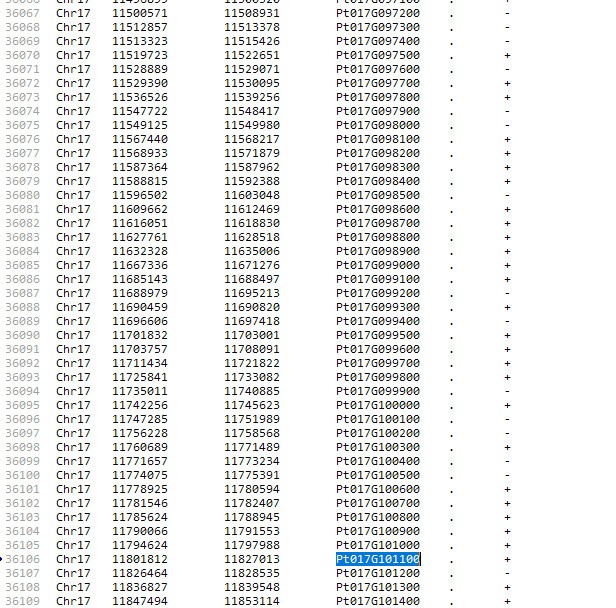

请老师指点,谢谢
请先 登录 后评论
- 1 关注
- 0 收藏,3778 浏览
- clcprince 提出于 2018-12-27 19:12
