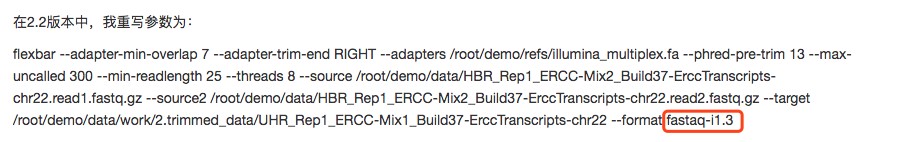
5 flexbar2.2版本去接头的过程中format不正确,报错文件格式不正确【课程RNAseq有参转录组数据分析】
flexbar版本: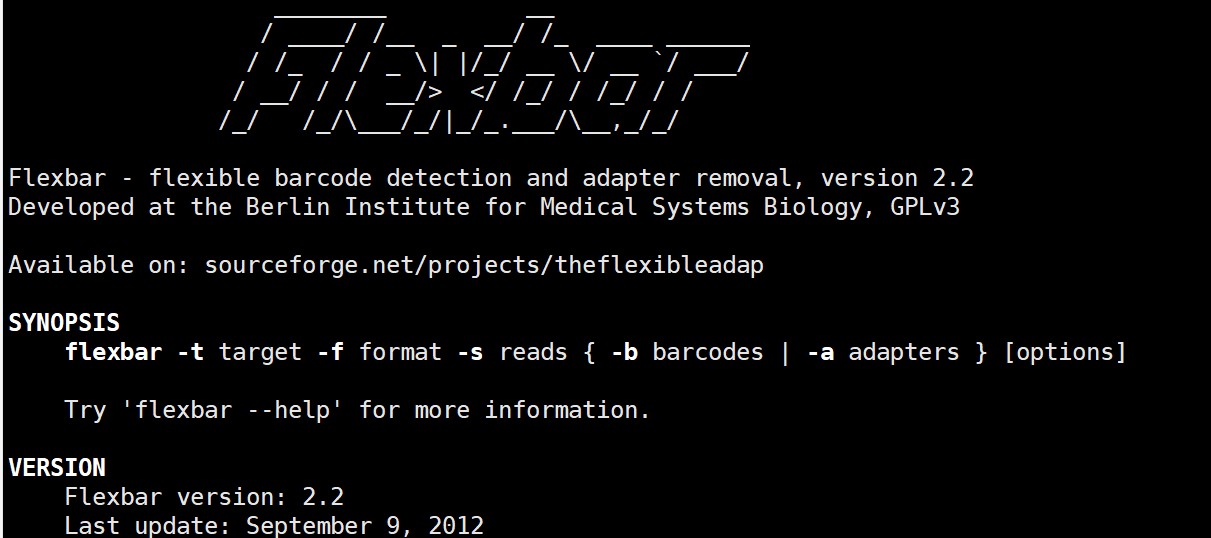
【课程RNAseq有参转录组数据分析】推荐的版本为2.5版本,参数如下:
flexbar
--adapter-min-overlap 7
--adapter-trim-end RIGHT
--adapters /var/data/work/refs/illumina_multiplex.fa
--pre-trim-left 13
--max-uncalled 300
--min-read-length 25
--threads 8
--reads /var/data/demo/data/UHR_Rep1_ERCC-Mix1_Build37-ErccTranscripts-chr22.read1.fastq.gz
--reads2 /var/data/demo/data/UHR_Rep1_ERCC-Mix1_Build37-ErccTranscripts-chr22.read2.fastq.gz
--target /var/data/work/2.trimmed_data/UHR_Rep1_ERCC-Mix1_Build37-ErccTranscripts-chr22
在2.2版本中,我重写参数为:
flexbar --adapter-min-overlap 7 --adapter-trim-end RIGHT --adapters /root/demo/refs/illumina_multiplex.fa --phred-pre-trim 13 --max-uncalled 300 --min-readlength 25 --threads 8 --source /root/demo/data/HBR_Rep1_ERCC-Mix2_Build37-ErccTranscripts-chr22.read1.fastq.gz --source2 /root/demo/data/HBR_Rep1_ERCC-Mix2_Build37-ErccTranscripts-chr22.read2.fastq.gz --target /root/demo/data/work/2.trimmed_data/UHR_Rep1_ERCC-Mix1_Build37-ErccTranscripts-chr22 --format fastaq-i1.3
报错为: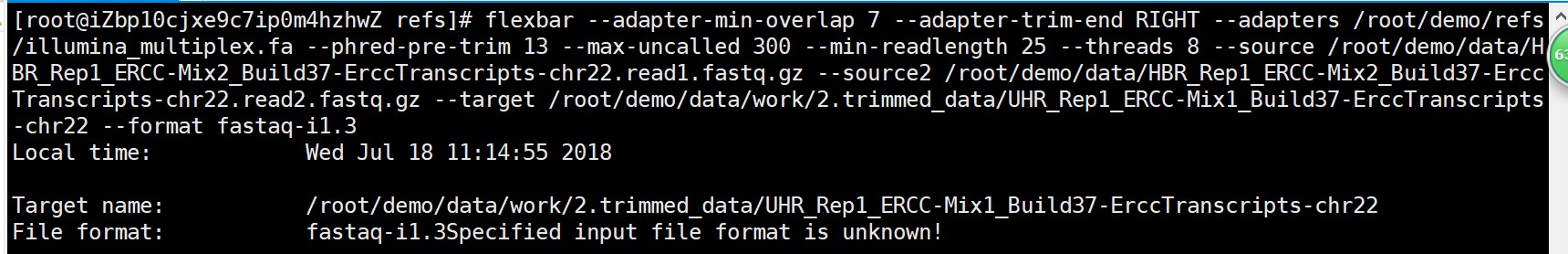
我的问题是:
1.我改写的参数是否正确,能够完成相同的任务么?
2.格式中format后面怎么写?
【2018年7月18号下午4点半补充问题】
1.对不起啊,存心大意了
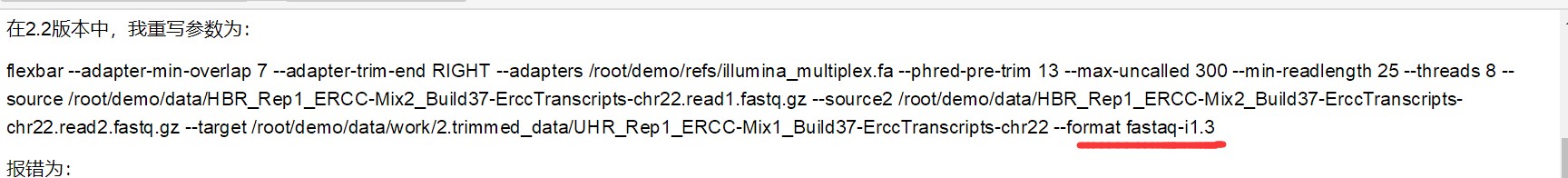
2.修改了之后是这个情况
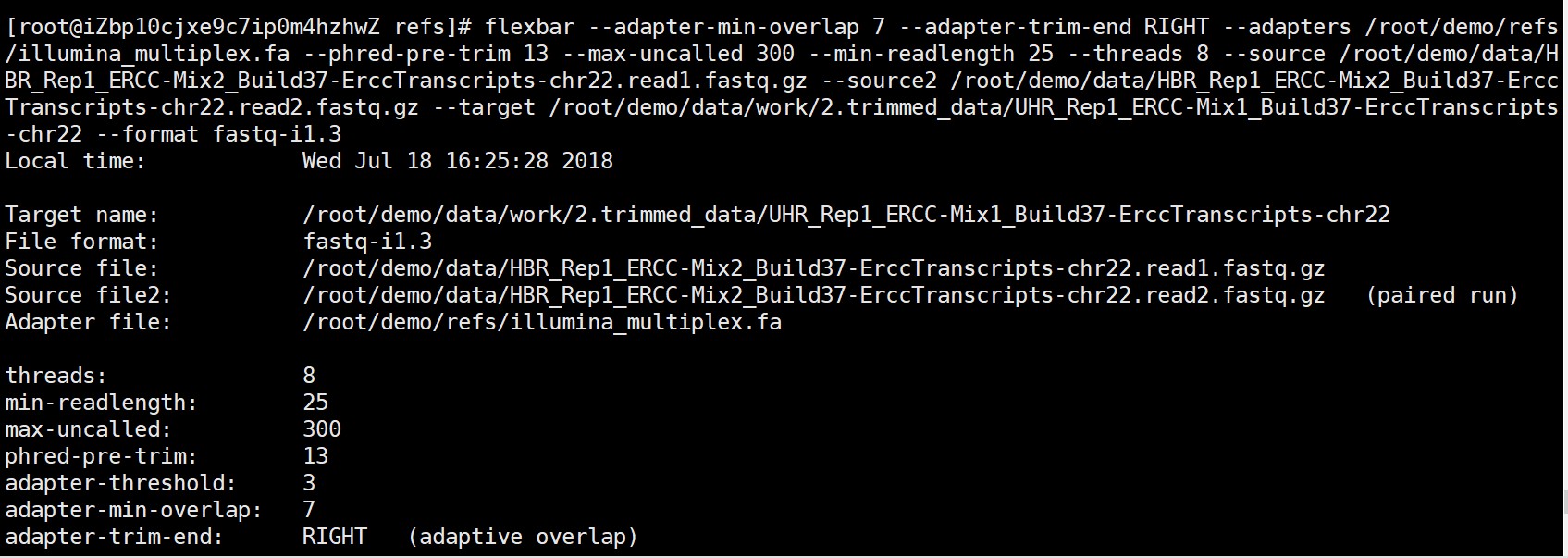
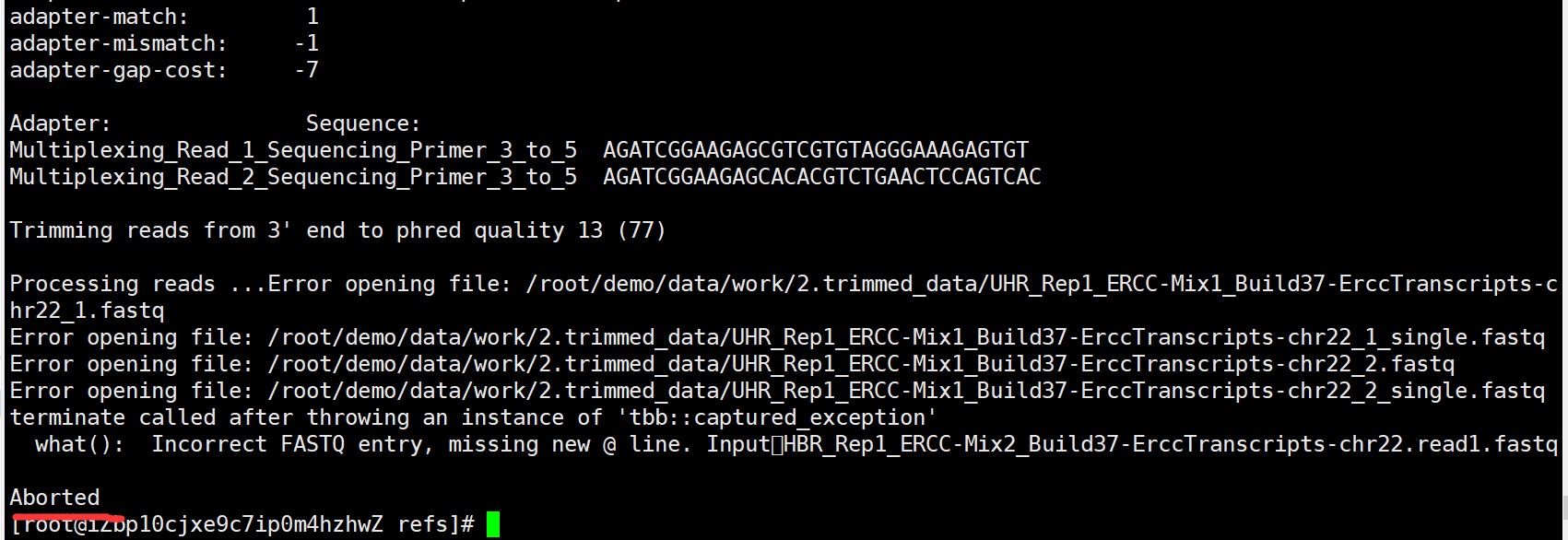
第二种情况才是我的本意,每次跑出来不对,不知道是不是参数忽略了。 麻烦各位老师了!
- 2 关注
- 0 收藏,5197 浏览
- Ideal 提出于 2018-07-18 11:16