没有基因组信息无法提取基因上游序列,我看你命令行输入的不是基因组序列吧;
5 如何修改perl脚本从cds文件中提取上游序列呢?(有些cds中的scaffold可能不足1500)
我想预测基因的顺式调控元件
但目前研究的物种并没有完整的染色体基因组信息
(下面是拟南芥的,完整的基因组信息)
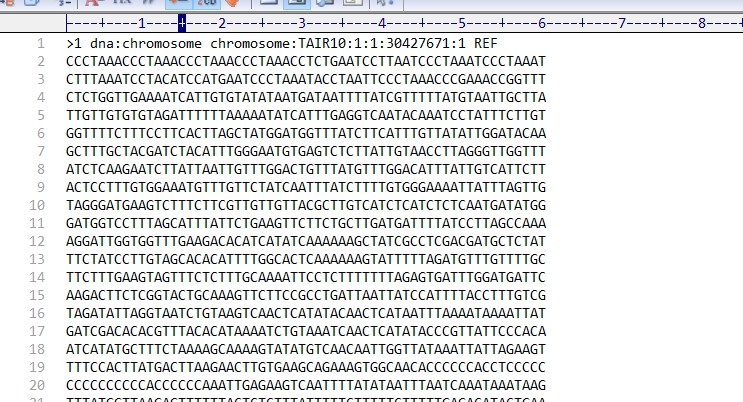 (下面是我研究的物种的,只有scaffold片段)
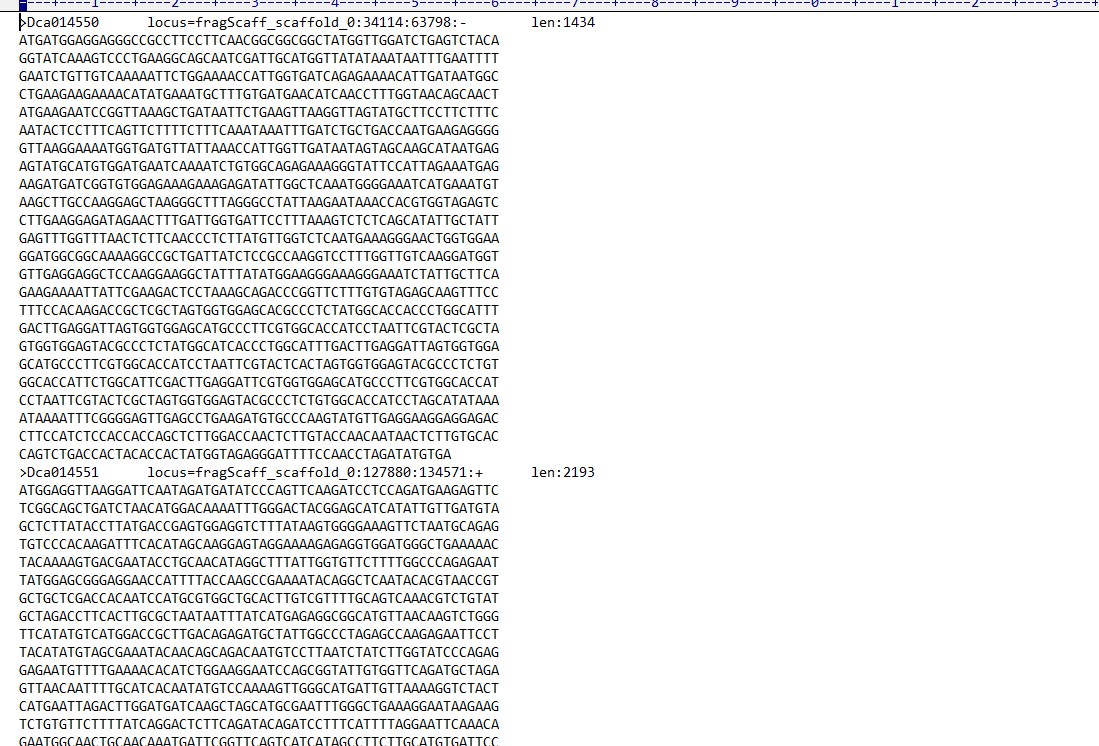
(下面是我研究的物种的,只有scaffold片段)
die "perl $0 <genome.fa> <weizhi.txt> <OUT> " unless(@ARGV==3 );
use Math::BigFloat;
use Bio::SeqIO;
use Bio::Seq;
$in = Bio::SeqIO -> new(-file => "$ARGV[0]",
-format => 'Fasta');
$out = Bio::SeqIO -> new(-file => ">$ARGV[2]",
-format => 'Fasta');
my %keep=() ;
open IN,"$ARGV[0]" or die "$!";
my%ref=();
while ( my $seq = $in->next_seq() ) {
my($id,$sequence,$desc)=($seq->id,$seq->seq,$seq->desc);
$ref{$id}=$seq;
}
$in->close();
open IN,"$ARGV[1]" or die "$!";
while (<IN>) {
chomp;
next if /^#/;
my @a= split /\t/;
my$seq=$ref{$a[1]};
print "$a[1]";
if( $a[4] eq "-" ){
$str= $a[3]+1;
$end=$a[3]+500;
my$seq_string=$seq->subseq($str,$end);
my$newseqobj1=Bio::Seq -> new(-seq => $seq_string,
-id => "$a[0]"
) ;
my$reseq = $newseqobj1 ->revcom();
$out->write_seq($reseq);
}elsif ( $a[4] eq "+" ){
$str= $a[2]-500;
$end=$a[2]-1;
my$seq_string=$seq->subseq($str,$end);
my$newseqobj1=Bio::Seq -> new(-seq => $seq_string,
-id => "$a[0]"
) ;
$out->write_seq($newseqobj1);
}
}
close (IN);
$in->close();
$out->close();

1 个回答
- 1 关注
- 0 收藏,4290 浏览
- Miracle.Joe╱ 提出于 2019-01-28 14:31
