没明白你的问题,物种分布指的是什么? 请贴图或者提供更多信息,才好回答。
2 个回答
你这个问题最好细节描述一下,或者给一张示例图什么的?不知道你指的是不是无参转录组里面 NR数据库的比对结果,结果一般是类似下面这张图片:
 (图片摘自:基于RNA-seq技术对乌鳢和斑鳢肝脏的转录组分析,刘凯,2015)
(图片摘自:基于RNA-seq技术对乌鳢和斑鳢肝脏的转录组分析,刘凯,2015)
首先确保本地建库过程中包含物种信息;
无参转录组分析,通过denovo组装获得unigenes,利用这些unigenes和NR数据库进行比对blast分析,获得同源基因及物种结果,之后绘图即可
blastx -outfmt 5
参数-outfmt 5可以输出xml,文件中hit_def能找到物种信息
补充评论七图片:
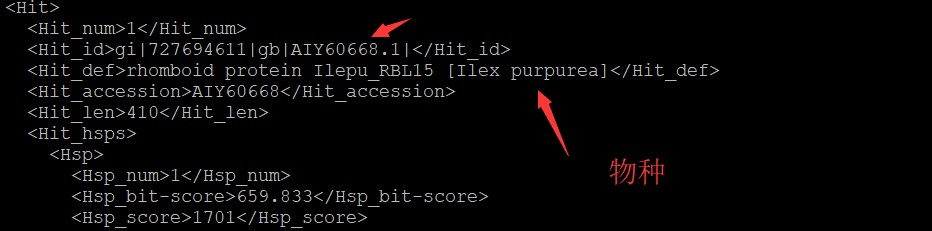
或者在-outfmt 6等(6、7、10、17)格式中控制输出内容:
-outfmt '6 ssciname'
请先 登录 后评论
- 3 关注
- 0 收藏,5103 浏览
- fangyue 提出于 2018-07-24 14:33
