10 (WGCNA) 已有基因模块分析结果,如何将其继续转换成可以用于cytoscape作图的文件?
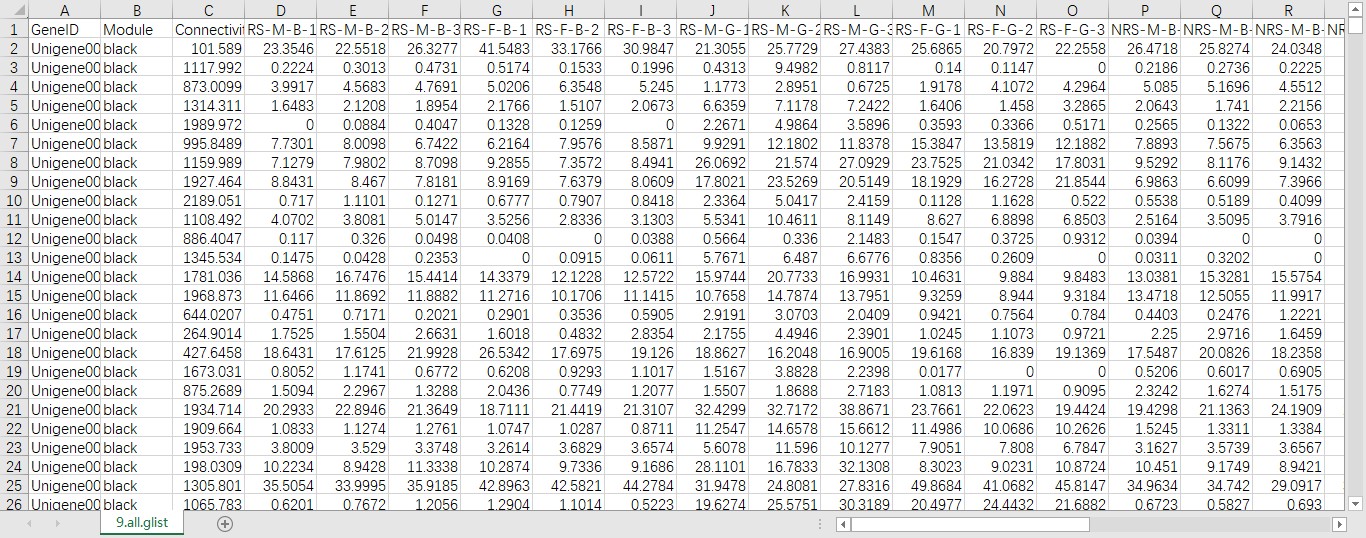 以上是模块基因总表的部分展示。
以上是模块基因总表的部分展示。
因为总基因数达10万多,设备不支持按课程从头做下来,但我已经有了模块基因总表,已经把基因分模块划分,且给出了连通性,如何进一步转换成cytoscape绘图的输入文件?
其他的文件也有,像是基因-模块对应关系列表、模块-模块相关性结果excel展示(包含所有模块两两之间特征值的相关性系数与p值)、基因-模块相关性结果excel展示(包含所有样本基因与模块特征值的相关系数与P值)、样本表达模式结果excel展示(包含模块特征值在各个样本中归一化的数值)。
其实就是WGCNA课程里将分析数据转换成cytoscape绘图文件之前的分析结果都以excel展示了,如何把这些excel结果继续转换成用于cytoscape绘图的文件?
先谢过各位老师!
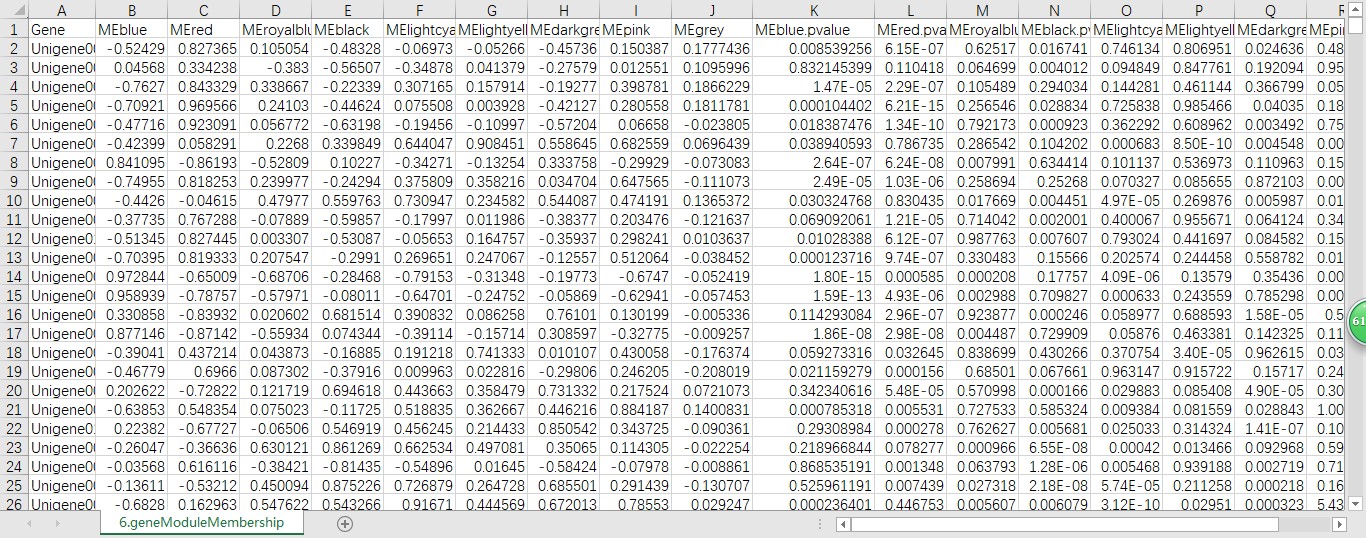
我这些文件里没有基因-基因的对应关系,我看到课程里有代码做基因-模块相关性分析热图(如下图),我这里有这个图相关的数据(包含所有样本基因与模块特征值的相关系数与P值),不知道可不可以用这个数据切入到原本的WGCNA分析过程里面?

请先 登录 后评论
- 2 关注
- 0 收藏,8278 浏览
- 听风 提出于 2019-05-13 11:40