你这个比较麻烦,蛋白ID和mRNA ID 不是一致的,导致脚本找不到对应的蛋白序列;
因为我的脚本认为 mRNAID和 cds pep 序列的ID一致;
#去除重复的hmmer搜索的转录本ID,多个转录本ID保留一个作为基因的代表,此步建议对脚本输出的文件手动筛选,挑选ID:
perl script/select_redundant_mRNA.pl mRNA2geneID.txt WRKY_domain_new_out_selected.txt WRKY_remove_redundant_IDlist.txt
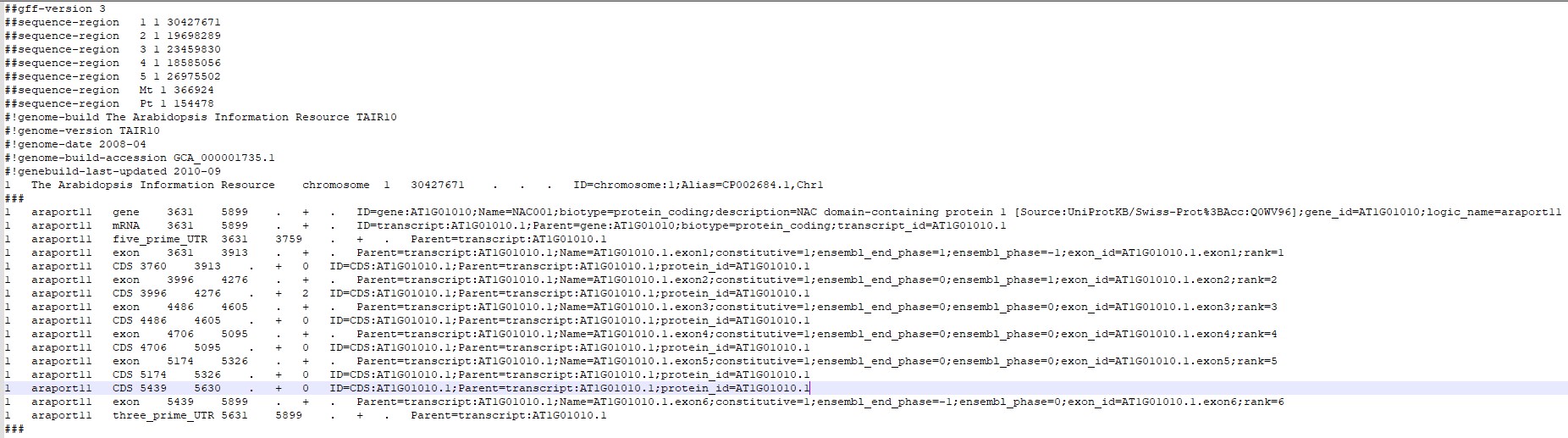
查看解答区的相似问题:老师的解答为:GFF文件可能不是标准的gff文件。我检查了自己的gff文件,第三列包含了gene,mRNA,cds等信息(标注2)。但是与拟南芥的gff文件相比,多了红色部分内容(标注1)。如图所示:
图一为我的gff文件信息
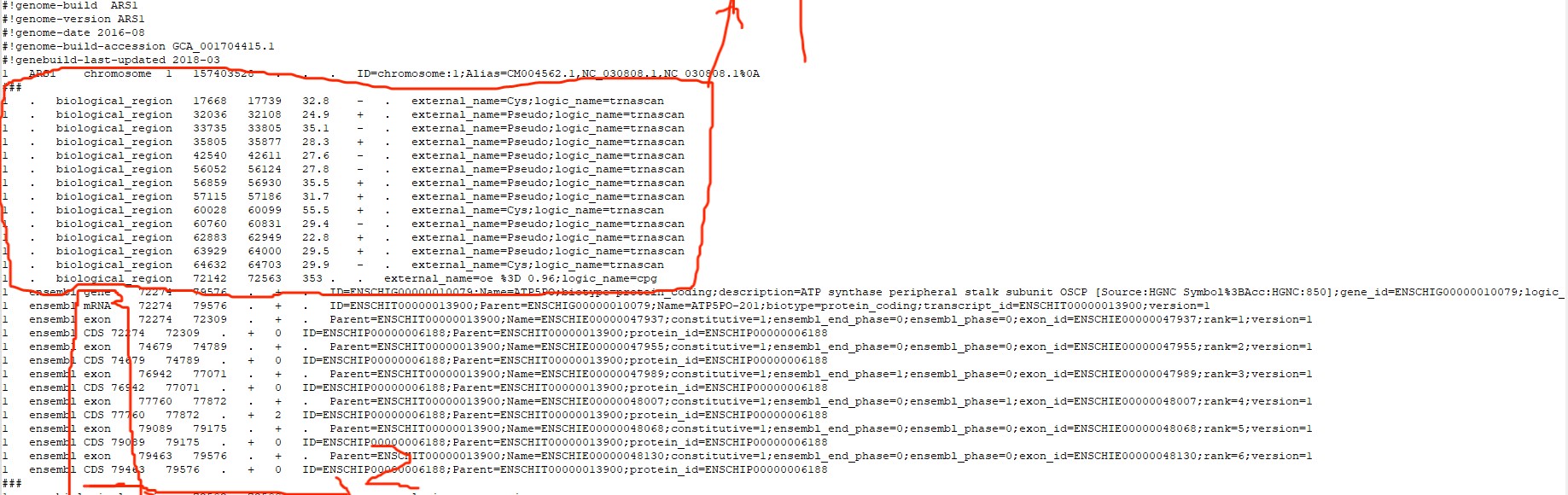
图二 拟南芥
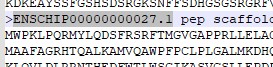
另外,我检查了蛋白ID和gffID,发现ID不一致。蛋白ID多了:.1。第一张为蛋白ID,第二张为gff ID。
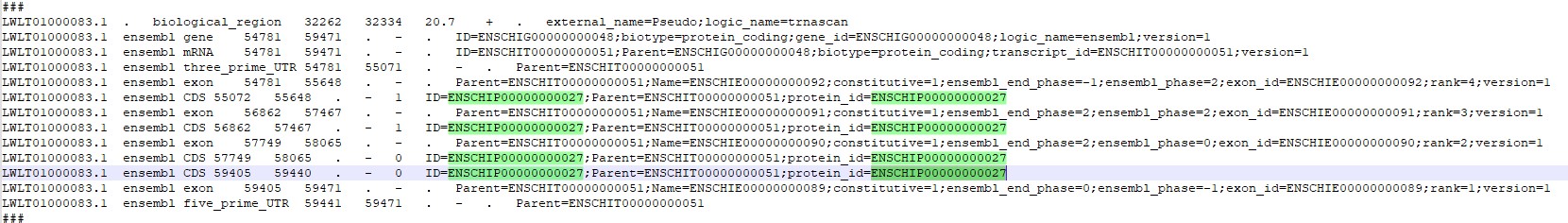
我的mRNA2geneID.txt 如下:
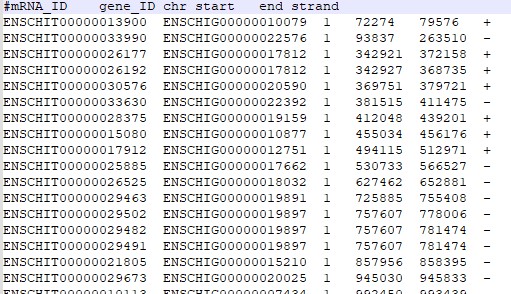 这是xxxx_domain_new_out_selected.txt截图:(gene 处连接为完整序列信息)
这是xxxx_domain_new_out_selected.txt截图:(gene 处连接为完整序列信息)


因此,为了去除重复,我应该怎么该我的文件?