10 利用samtools将sam文件转化为bam文件时,sam文件报错
运行命令:
$samtools sort -@ 4 -O bam -o RNA.bam RNA.sam
报错:
[E::sam_hdr_create] Invalid header line: must start with @HD/@SQ/@RG/@PG/@CO
samtools sort: failed to read header from "RNA.sam"
RNA.sam文件行首:
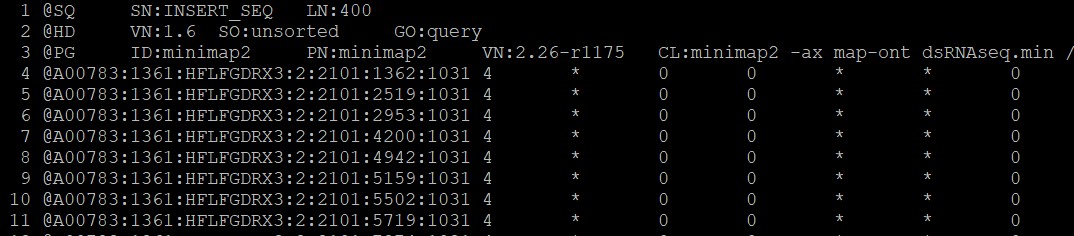
请先 登录 后评论
- 1 关注
- 0 收藏,2566 浏览
- rzx 提出于 2023-10-11 15:33