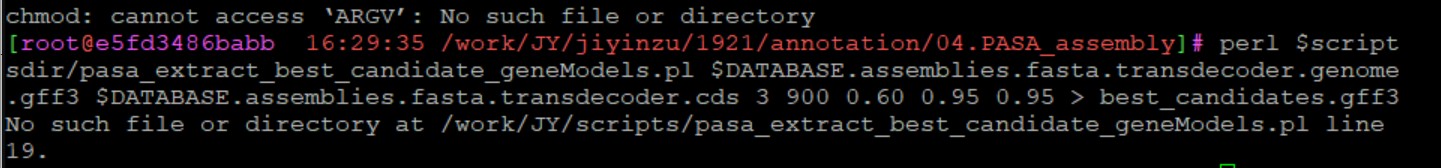
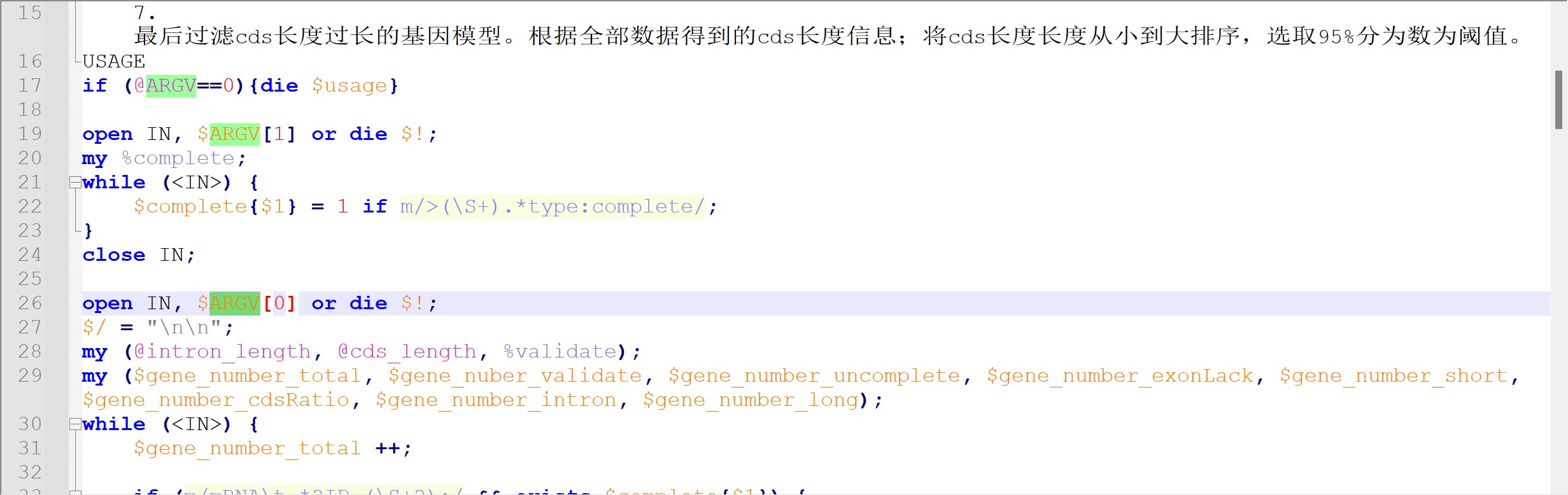
5 进行基因注释过程中运行这个脚本perl $scriptsdir/pasa_extract_best_candidate_geneModels.pl $DATABASE.assemblies.fasta.transdecoder.genome.gff3 $DATABASE.assemblies.fasta.transdecoder.cds 3 900 0.60 0.95 0.95 > best_candidates.gff3出现错误。希望老师解答,谢谢
- 1 关注
- 0 收藏,1677 浏览
- Jolly 提出于 2024-01-25 20:43