用的是我们的docker镜像分析的吗?如果不是那就是软件环境配置问题,
建议用我们提供的docker镜像分析;
计算机使用的是公共集群,使用conda配置的环境,使用conda安装的qualimap。
使用sbatch提交命令
运行命令如下,因为字数限制将软件路径和文件路径都省略了:
#!/bin/bash
conda activate mapping
ls ${bwadir}/*.sorted.dedup.bam | while read id
do
name=${id##*/}
name=${name%%.*}
#1.比对率分析
$samtools flagstat ${bwadir}/${name}.sorted.dedup.bam > ${bwa_qcdir}/${name}.map.stat.txt
#2.片段inner size,片段选择是否异常
$picard CollectInsertSizeMetrics -Xmx4g \
I=${bwadir}/${name}.sorted.dedup.bam \
O=${bwa_qcdir}/${name}.insert_size_metrics.txt \
H=${bwa_qcdir}/${name}.insert_size_histogram.pdf \
M=0.5
#3.测序深度与覆盖度统计
$qualimap bamqc --java-mem-size=2G -nt 8 -nr 10000 -bam ${bwadir}/${name}.sorted.dedup.bam -outfile report.pdf -outformat PDF -outdir ${bwa_qcdir}/${name}.depth_and_coverage_report
done
if [ $? -eq 0 ]; then
touch ${bwa_qcdir}/ok.cucumber_bwa.qc.status
else
touch ${bwa_qcdir}/failed.cucumber_bwa.qc.status
fi
最后运行的时候 #1.比对率分析和#2.片段inner size,片段选择是否异常这两个能正常运行成功,但是#3.测序深度与覆盖度统计这一部分qualimap出现报错
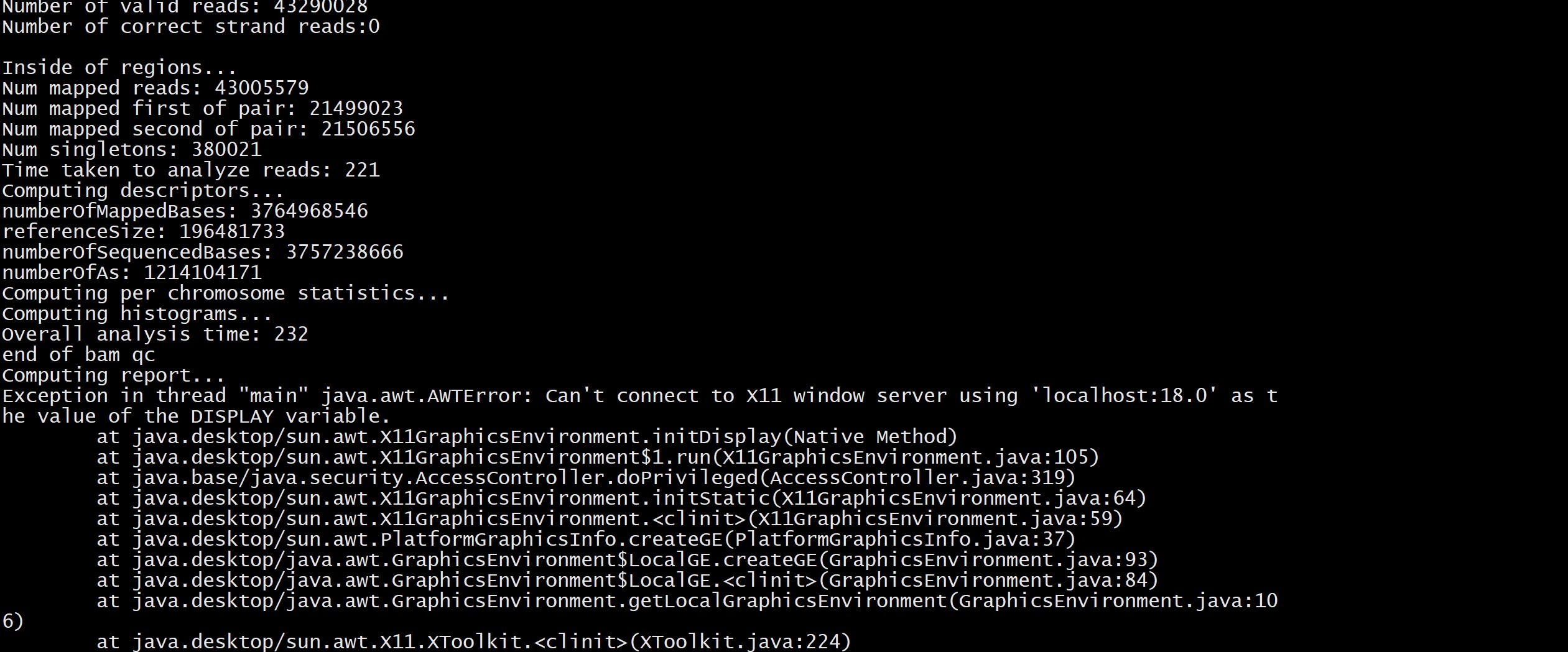
后续在网上搜了一下需在~/.bashrc 环境种输入这段修改JAVA的环境设置
export JAVA_OPTS=-Djava.awt.headless=true
但是这样也还是不行,还是显示上面报错。
请老师们帮忙解答一下怎么解决!