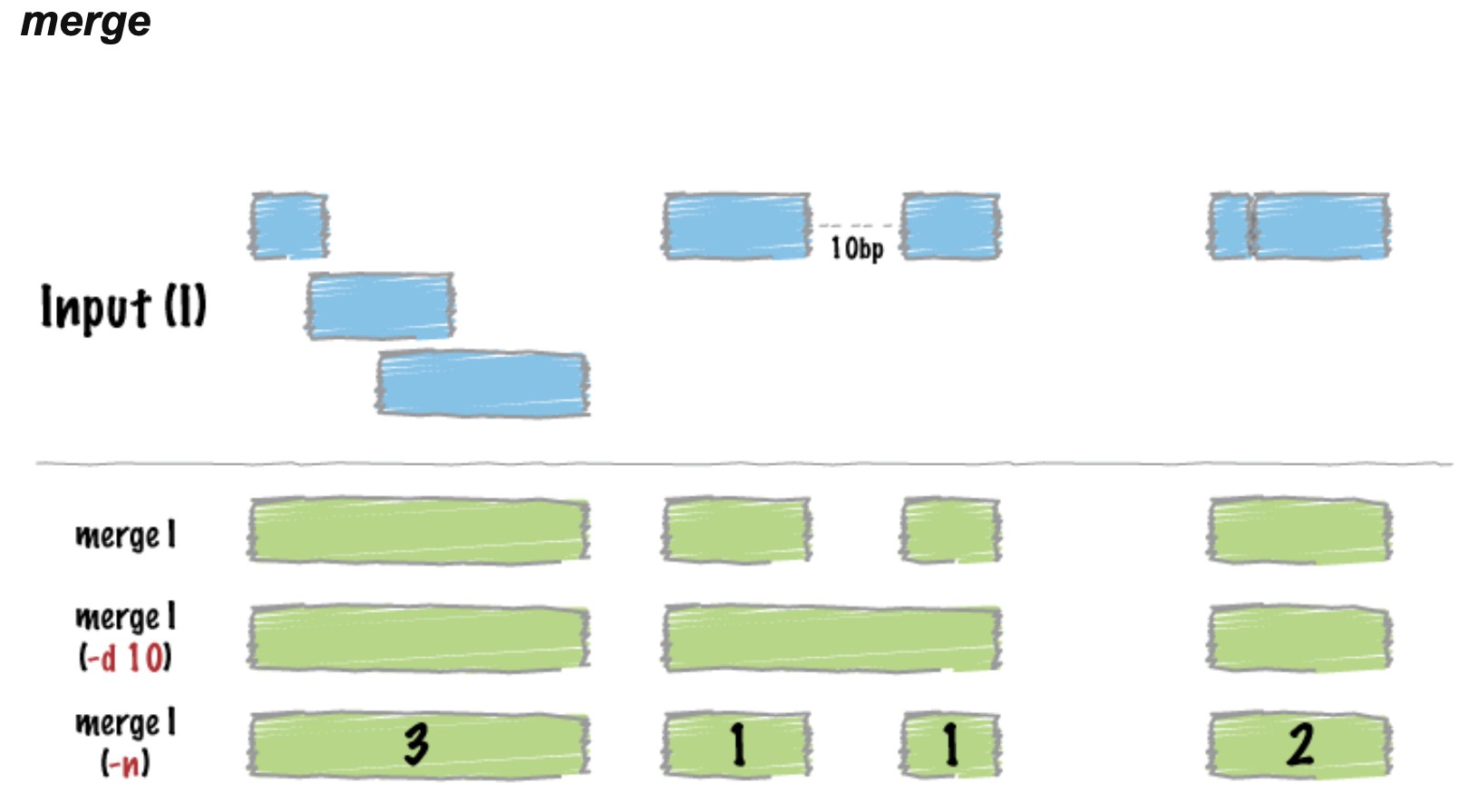
这一步不是在合并p值,是在合并snp位点和上下游10kb区域的位置信息,需要做
GWAS做关联区域分析时,根据p值获取关联区域中,为什么要用如下语句以合并P值(此语句是2023.4.10日添加的) cat pvalue.bed region.bed |sort -k1,1 -k2,2n >all.region.bed 合并P值在获取关联区域的作用是什么,可否像之前录像讲的那样不用这个语句吗?
2 个回答
- 2 关注
- 0 收藏,236 浏览
- John 提出于 2025-03-12 15:34
相似问题
- 请问老师,为什么显示找不到文件呢,已经尝试过重新传输文件 1 回答
- 老师您好,针对这个问题https://www.omicsclass.com/question/7725。你们提到:你这个数据里面是不是P值有0的情况,取了对数就无穷大了,导致出问题。但是,我发现里面没有P值为0的情况,那是不是脚本gwas_manhattan_plot.r有问题呢?需要修改gwas_manhattan_plot.r脚本的什么地方才可以解决顶部只能画半圆的问题呢?还有,我发现P-Value的原始文件有NaN的情况,是什么原因呢?是否对GWAS有影响呢? 1 回答
- GWAS分析发现的问题如下:第一,过滤水稻vcf的RawData时(群体有176份),我选择的minGQ值为20(同时要移除InDel),结果得出过滤出来的SNPs数量不足1000,但是我查过一些资料,GQ值如果群体数量较少值不得小于20。请问遇到这种情况如何处理,可否将最小GQ按照课程的脚本设置为0呢。 第二,你们的GWAS课程提供的计算BLUP的脚本是否在不同物种之间通用 第三,做GWAS时,基因型填充对后续的分析有什么意义。 第四,你们GWAS课程有没有计算遗传力相关的脚本。 1 回答
- 我在win11本地GWAS分析时,进行plink。结果报错了(Segmentation fault (Core dumped)),请问各位如何解决 2 回答
- 采用你们课程的:Rscript $scriptdir/gwas_manhattan_plot.r 以及Rscript $scriptdir/qq_plot.r制作Manhattan图和QQ图后,Manhattan图最高的SNP仅仅显示半圆(如下图),请问如何修复(就是修改可视化Manhattan图的纵坐标) 2 回答