内置的 基因集数据库用的R包是这个:msigdbr 说明:https://igordot.github.io/msigdbr/articles/msigdbr-intro.html
常见基因集:
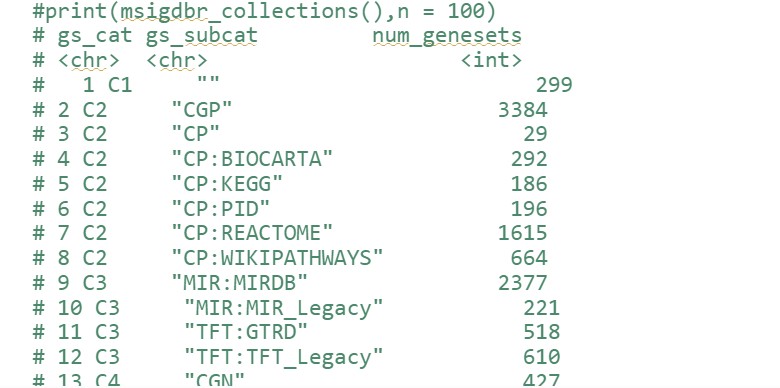
| gs_cat | gs_subcat | num_genesets |
| C1 | 299 | |
| C2 | CGP | 3384 |
| C2 | CP | 29 |
| C2 | CP:BIOCARTA | 292 |
| C2 | CP:KEGG | 186 |
| C2 | CP:PID | 196 |
| C2 | CP:REACTOME | 1615 |
| C2 | CP:WIKIPATHWAYS | 664 |
| C3 | MIR:MIRDB | 2377 |
| C3 | MIR:MIR_Legacy | 221 |
| C3 | TFT:GTRD | 518 |
| C3 | TFT:TFT_Legacy | 610 |
| C4 | CGN | 427 |
| C4 | CM | 431 |
| C5 | GO:BP | 7658 |
| C5 | GO:CC | 1006 |
| C5 | GO:MF | 1738 |
| C5 | HPO | 5071 |
| C6 | “” | 189 |
| C7 | IMMUNESIGDB | 4872 |
| C7 | VAX | 347 |
| C8 | “” | 700 |
| H | “” | 50 |
如果 没有自己关注基因集 可以按照示例数据自己准备基因集文件;