还是用之前的脚本合并:示例脚本在: 03.seurat_cluster.sh
#合并数据
Rscript $scripts/merge_seurat_obj.r -i CD4.added.celltype.rds CD8.added.celltype.rds -p all.sample.merged
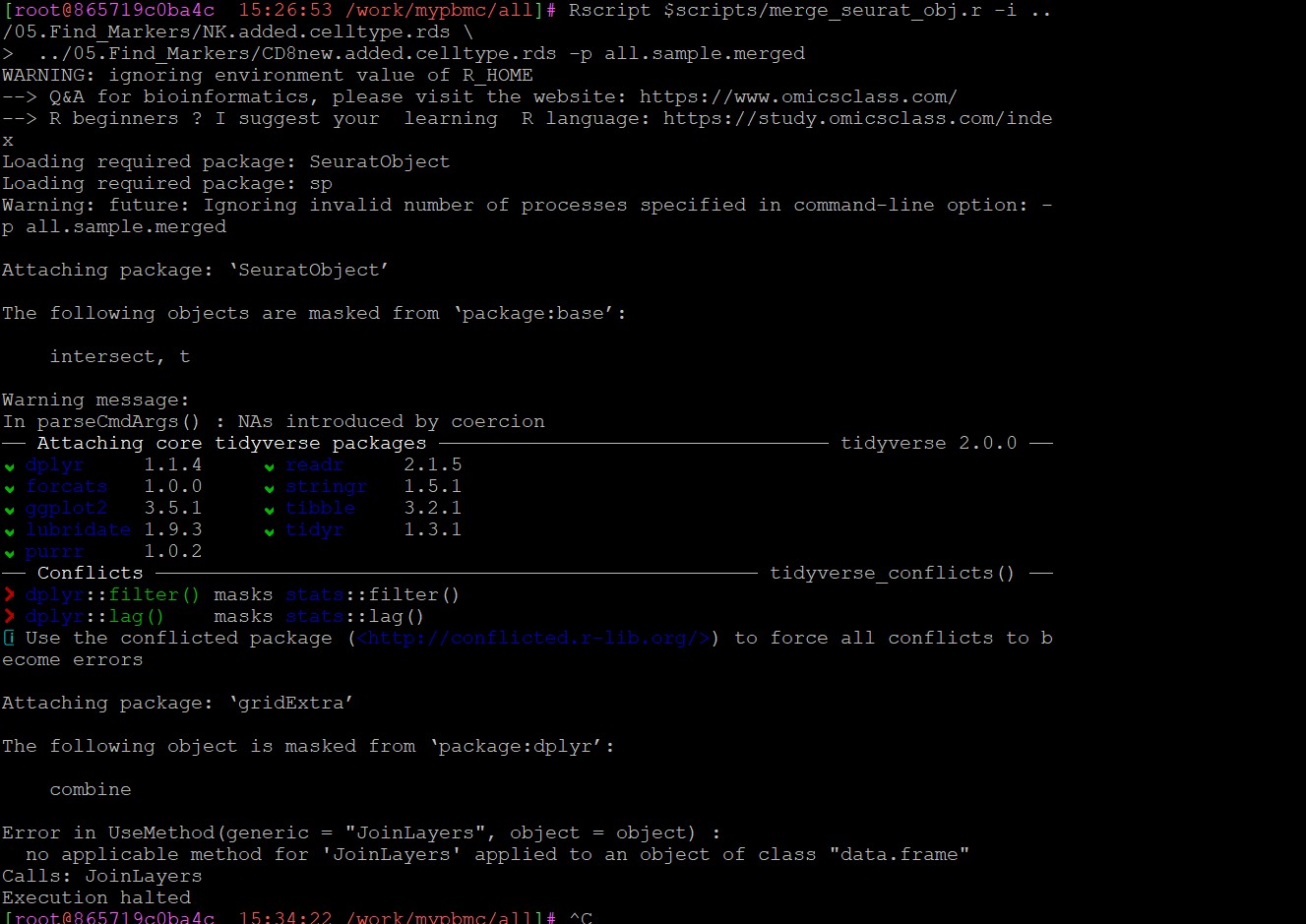
我把CD8T和NK细胞取子集后进行了重聚类,resolution都是0.2,然后生成了CD8new.added.celltype.rds和NK.added.cellytype.rds两个文件,现在我要把这两个文件,但总是提示报错。
CD8new.added.celltype.rds的meta文件:CD8.added.celltype.metadata.tsv
NK.added.cellytype.rds的meta文件:NK.added.celltype.metadata.tsv
我的代码:Rscript $scripts/merge_seurat_obj.r -i ../05.Find_Markers/NK.added.celltype.rds \
> ../05.Find_Markers/CD8new.added.celltype.rds -p all.sample.merged
我的报错: