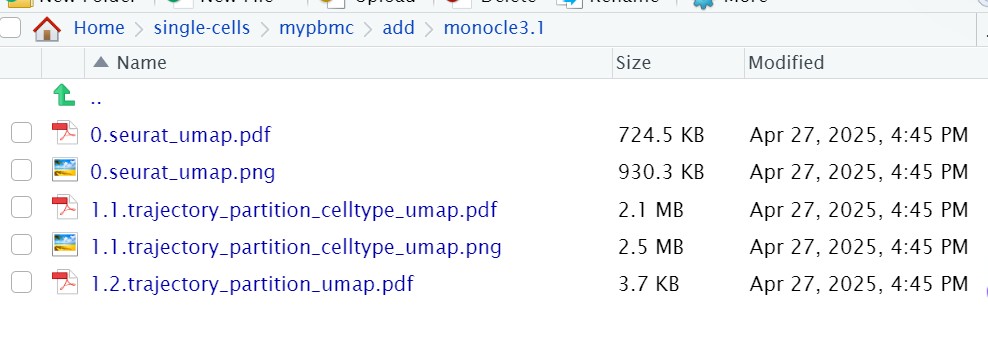
 老师,图生成到这里就停了
老师,图生成到这里就停了
使用monocle3进行轨迹分析报错
使用单细胞转录组学课程中的代码使用monocle3进行轨迹分析报错。
这是我的代码: Rscript $scripts/cell_trajectory_monocle3.r --rds ../05.Find_Markers/CD4.added.celltype.rds \--use.seurat.umap --groupby mycelltype -o monocle3.1 --cpu 4
这是CD4.added.celltype.rds所对应的meta文件:
CD4.added.celltype.metadata.tsv
这是报错: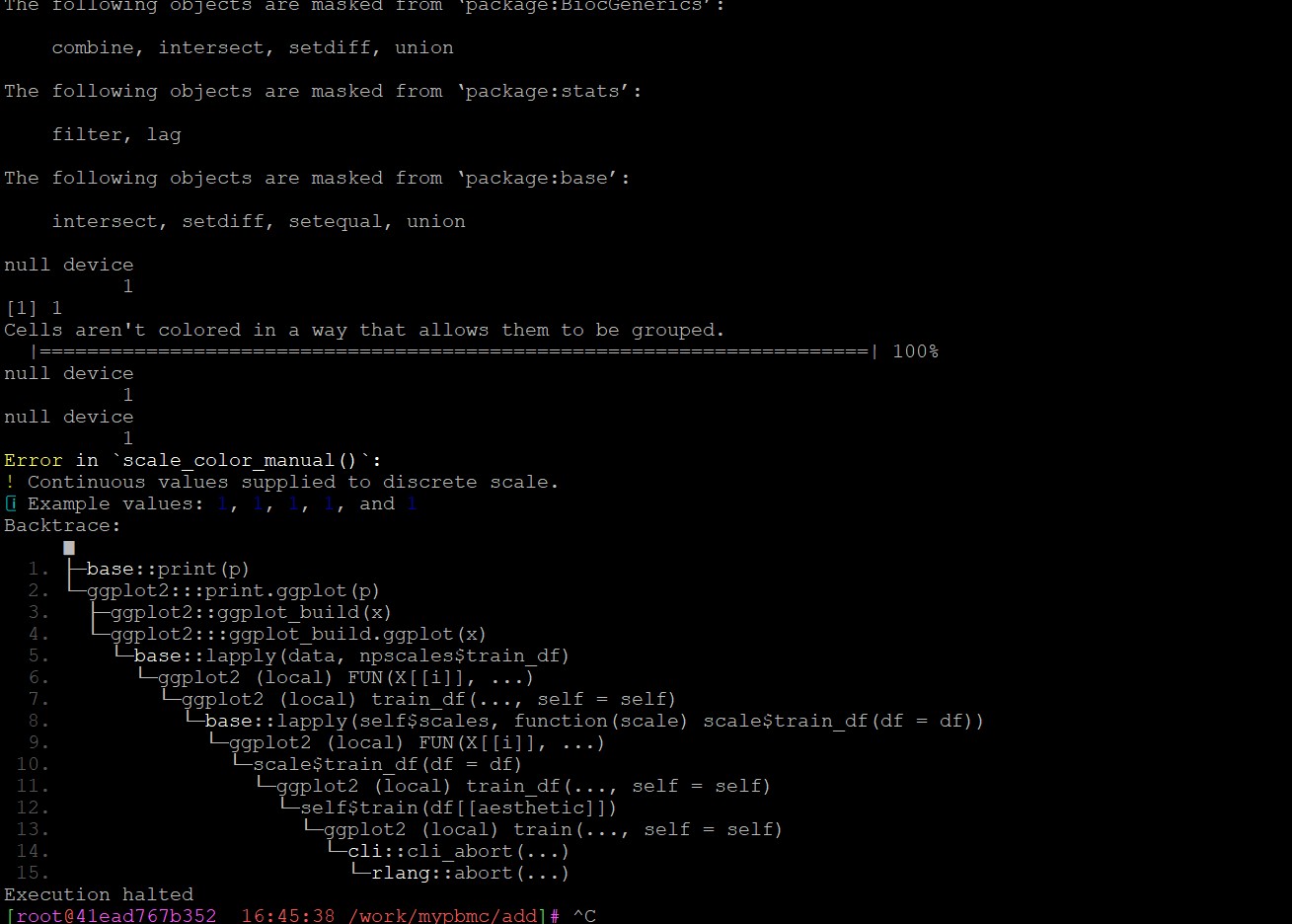
按照老师的要求修改代码之后依然报错,而且报同样的错:
我修改的代码:
报错: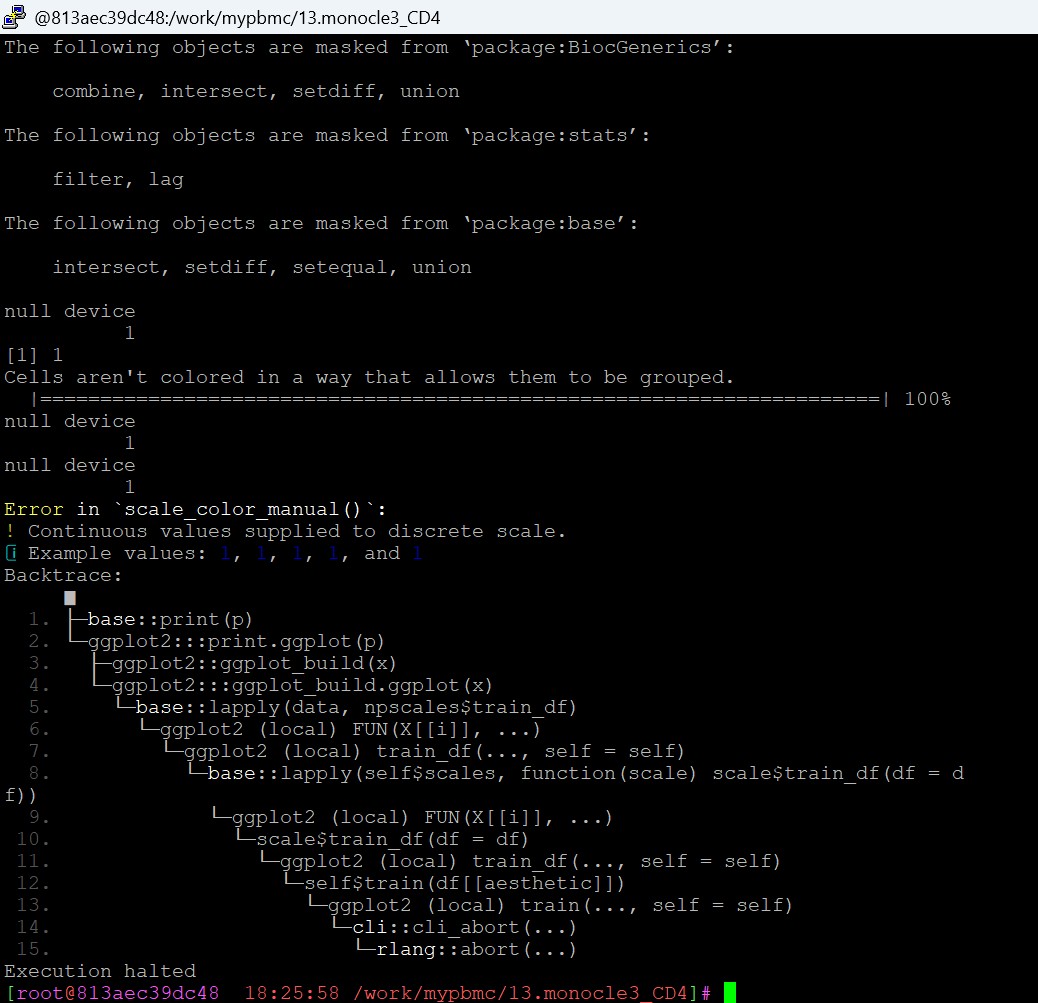
出图: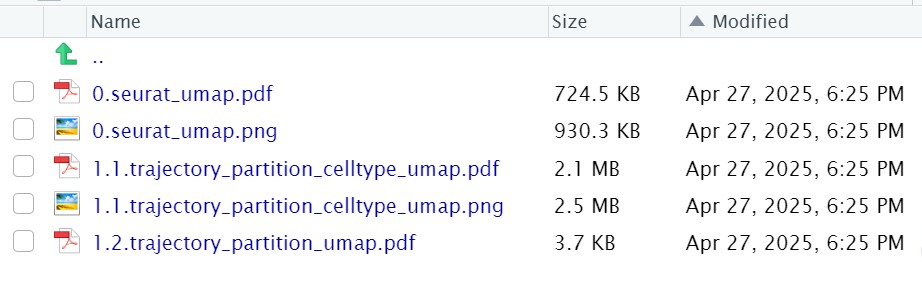
请先 登录 后评论
2 个回答
- 2 关注
- 0 收藏,83 浏览
- Zzz 提出于 2天前
相似问题
- 单细胞测序的细胞通讯分析 cellchat 1 回答
- 文件夹权限受限,无法删除 1 回答
- 单细胞测序数据两个子集合并 1 回答
- 如何合并两个单细胞测序中的两个子集? 1 回答
- 使用monocle3进行轨迹分析报错 0 回答

